中国幽门螺杆菌cagA克隆和基因序列分析
作者:钟 平 吴昆详 徐帆洪 许春娣 徐家裕
单位:钟 平 吴昆详 徐帆洪 上海生物制品研究所诊断试剂研究室 上海市 200052; 许春娣 徐家裕 上海瑞金医院小儿内科 上海市 200025
关键词:幽门螺杆菌感染;细胞毒素相关基因A;基因克隆;cagA序列分析
世界华人消化杂志990810 中国图书馆分类号 R573
摘 要
目的 了解和分析中国幽门螺杆菌(Helicobacter pylori, Hp)菌株细胞毒素相关基因A(cagA)的核苷酸序列特性.
方法 从两位患胃溃疡儿童的胃粘膜中分离两株Hp菌株,用PCR方法扩增cagA基因的5端和3端的两个片段并作克隆和序列分析.
, http://www.100md.com
结果 两株中国Hp菌株cagA基因之间的同源性为94%~95%. 中国Hp菌株与国外菌株相同区段比较,5端的同源性为89%~92%,3端的同源性为69%.
结论 中国Hp菌株与国外Hp菌株cagA基因5端序列部分区域的同源性较好,可以作为分子检测Hp感染的靶基因序列,而cagA基因3端序列部分区域的同源性较差,可以作为Hp分子流行病学研究的靶基因序列.
Cloning and characterization of Hp cagA gene sequences derived from Chinese strains
ZHONG Ping1, WU Kun-Xiang1, XU Fan-Hong1, XU Chun-Di2 and XU Jia-Yu2
, 百拇医药
1Laboratory of Diagnostic Reagents, Shanghai Institute of Biological Products, Shanghai 200052, China
2Shanghai Ruijin Hospital, Shanghai 200025, China
Subject headings Helicobacter pylori infection; cagA; gene cloning; cagA gene; sequencing
Abstract
AIM To investigate the sequence characterization of Hp cagA gene from Chinese strains.
, 百拇医药
METHODS Two Hp strains derived from two Chinese pediatric patients with gastric ulcer were isolated. Two DNA fragments of Hp cagA gene 5 and 3 regions of each strain were PCR-amplified, cloned and sequenced.
RESULTS The nucleotide homology between two Chinese Hp strains was 94%. However, the nucleotide homology of cagA gene 5 and 3 regions between Chinese and American strains and between Chinese and Italian strains were 89% and 69%-71%, respectively.
, 百拇医药
CONCLUSION The nucleotide sequences of Hp cagA gene 5 region among the strains showed a better homology, this region could be chosen as a target gene for molecular diagnosis of Hp infection, while that of cagA gene 3 region between Chinese and American strains or between Chinese and Italian strains showed a poorer homology, this region could be chosen as a target gene for Hp molecular epidemiological study.
0 引言
幽门螺杆菌(Helicobacter pylori, Hp)是消化性溃疡和慢性胃炎的主要致病因子,并与胃癌的发生有密切关系. 目前国外已经发表了近10个Hp菌株cagA基因的全序列,然而中国Hp菌株的cagA基因的序列还未见报道. 为此,我们从中国患胃溃疡儿童胃粘膜中分离了两株Hp,进行分子克隆和分析了它们的cagA基因中的两个片段的核苷酸序列,为中国分子诊断Hp感染和进行Hp的分子流行病学研究奠定基础.
, 百拇医药
1 材料和方法
1.1 材料 胃粘膜组织取自于上海第二医科大学瑞金医院儿科两位胃溃疡儿童. Hp分离培养基:培养基成分为:Campylobacter Agar Base 43g/L(OXOID),奈啶酸20mg/L(Sigma),TTC 40mg/L (F.M.K.),万古霉素6mg/L(Sigma),两性霉素B2mg/L(Sigma),100mL/L新鲜羊血. pMAL-C2购自于美国Biolabs公司,pSP72和大肠杆菌JM109由本所分子生物学研究室提供. 限制性内切酶和T4 DNA连接酶(Boehringer Mannheim), Taq plusⅡ DNA聚合酶(上海Sangon公司),T7测序试剂盒(Pharmacia),α-35S-dCTP(Amersham). Hp cagA 5端核苷酸序列片段的PCR扩增引物(cagA基因157nt~504nt):P1(上游引物):5-CCGGAGAATTCGATAACAGGCAAGCTTTTGAGG-3;P2(下游引物):5-GCCTGCAGTAATCGAAAAGA_TTGTTTGGCAG-3;Hp cagA 3端核苷酸序列片段的PCR扩增引物(cagA基因2269nt~2877nt):P3(上游引物):5-GCGGATCCAGCAAGGTAACGCAAGC-3;P4(下游引物):5-GCGTCGACTTATCGCCCTACTTCACTGA_GATC-3. 引物由上海Sangon公司合成.
, 百拇医药
1.2 方法 内镜下应用常规方法在胃窦部炎症明显处铗取胃粘膜组织块,接种于选择培养基平板,置37℃微氧环境(50mL/L O2,70mL/L CO2,80mL/L H2和800mL/L N2)下培养5d. Hp基因组DNA的制备参照文献[1]中的方法. PCR扩增cagA基因5端和3端片段:PCR反应体积为50μL,其中包括主要试剂0.3μmol/L引物,1.5mmol/L Mg2+,0.5U Taq DNA聚合酶. PCR反应条件:94℃ 4min预变性;94℃ 1min,55℃ 1min,72℃ 1min,共35个循环;72℃延伸10min. DNA片段纯化、质粒连接和细菌转化参照文献[2]方法进行. DNA序列测定和分析采用Sanger双脱氧链终止法测序. 采用电脑DNA分析辅助软件对所测得的DNA序列进行同源性分析.
2 结果
, http://www.100md.com
2.1 PCR扩增cagA基因片段 5端片段PCR片段扩增产物长度为340bp,与预期的核苷酸长度相符合;3端片段PCR扩增产物的长度为700bp,比预期的长度604bp长.
2.2 测序质粒的构建 PCR扩增的Hp cagA基因5端片段和3端片段分别经EcoRⅠ, PstⅠ和BamHⅠ, SalⅠ双酶切反应后分别定向插入于载体pMAL-C2和pSP72中,得到四个测序重组质粒如pMA1, pMA2, pSB1和pSB2. 它们的双酶切反应后的片段与预期的核苷酸长度结果相符合.
2.3 Hp cagA基因5端和3端部分核苷酸序列和同源性分析 Hp cagA基因5端部分核苷酸序列中,两株中国Hp菌株(SH21和SH9)之间的最大同源性为94%~95%,它们与美国和意大利Hp菌株核苷酸之间的最大同源性为89%~92%(图1). Hp cagA基因3端部分核苷酸序列中,两株中国Hp菌株(SH21和SH9)之间的最大同源性为94%,而它们与美国和意大利Hp菌株核苷酸之间的最大同源性为69%(图2).
, http://www.100md.com
10 20 30 40 50 60
Hp(A) GATAACAGGC AAGCTTTTGA GGGAATCTCG CAATAAAGGG AAGAATACTC CAATAAAGCG
Hp(I) ********** ********** A********* ****T***** ********** **********
Hp(21) ********** ********** **A******* ***CT***** ********G* **********
Hp(9) ********** ********** **A******* ***CT***** ********G* **********
70 80 90 100 110 120
, 百拇医药
Hp(A) ATCAAAAATC CTACCAAAAA GAATCAGTAT TTTTCAGACT TTATCAATAA GAGCAATGAT
Hp(I) ********** ********** ********** ********** ********** **********
Hp(21) ********** *C******** ********** ********** ********** **********
Hp(9) ********** *CG******* ********** ********** ********** **********
130 140 150 160 170 180
Hp(A) TTAATCAAGA AAGACAATCT CATTGTCGTG GAATCTTCCA CAAAGAGCTT TCAGAAATTT
, http://www.100md.com
Hp(I) ********C* ********** *****AT**A ********** ********** **********
Hp(21) **G*****C* ********** *****CT**A **T******G T*G******* *A********
Hp(9) **G*****C* ********** *****CT**A **T******G T*G*T***** *A********
190 200 210 220 230 240
Hp(A) GGGGATCAGC GTTACCGAAT TTTCACAAGT TGGGTGTCCC ATCAAAACGA TCCGTCTAAA
Hp(I) ********** ********** ********** ********** ********** **********
, 百拇医药
Hp(21) ********** ******A*** ***T**G*** ********** T******A** **********
Hp(9) ********** ******A*** ***T**G*** ********** T******A** **********
250 260 270 280 290 最大同源性
Hp(A) ATCAACACCC GATGCATCCG AAATTTTATG GAACATACCA TACAACCCCC TATCCCT
Hp(I) ********** ***CG***** ********** ***A***T** ********** *****T* 96%
Hp(21) ********** A*CAA***** ********** ***A***TT* ********** ****T** 90%92%
, 百拇医药
Hp(9) ********** A*CAA****A ********** ***A***T** ********** ****T** 89%90%95%
图1 幽门螺杆菌cagA基因5端片段核苷酸同源性分析. Hp(A):参考文献[4]菌株;Hp(I):参考文献[3]菌株;Hp(21)和Hp(9):临床分离菌株.
*表示相同核苷酸.
10 20 30 40 50 60
Hp(A) TTCAGCAAGG TAACGCAAGC AAAAAGCGAC CAAGAAAATT CCATTAAAGA TGTGATCATC
Hp(I) ********** ********** ********** *TT******* **G******* **********
, 百拇医药
Hp(21) ********** ********** ********** *TT******* *******G** *********T
Hp(9) ********** ********** ********** *TT******* *******G** **********
70 80 90 100 110 120
Hp(A) AATCAAAAGA TAACGGATAA AGTTGATGAA-CTCAATCAA GCGGTATCAG TGGCTAAAAT
Hp(I) *********G ********** *******A*T-********* ********** ********GC
Hp(21) ********** ********** ******C-** T******** G **T******* A*A*****T*
, 百拇医药
Hp(9) ********** ********** ******A-** T******** G **T**G**** A*A*****T*
130 140 150 160 170 180
Hp(A) AGC-GTGCGA TTTCAGTGGG GTAGAGCAAG CGTTAGCCGA TCTCAAAAAT TTCTCAAAGG
Hp(I) *A*-*G*T** *******A** ********** *CC******* ********** **********
Hp(21) A*A-G***** *******AA* ********** CC******** *********C **G***TT**
Hp(9) *A*A*-**** *******AA* ********** *CC******* A******GC* **G***TT**
, 百拇医药
190 200 210 220 230 240
Hp(A) AGCAATTGGC TCAACAAGCT CAAAAAAATG AAAGTT-TCA ATGTTGGA- - - - - - TCTGA
Hp(I) ********** C********* ********** **-**CC*** ***C*A**AA AAAA******
Hp(21) *T*--****---**-**----**-----*-----*---* -**----*------***CC*
Hp(9) *T*--****---**-**----**-----*-----*---* -**----*------***-*-
250 260 270 280 290 300
, 百拇医药
Hp(A) ATATACCAAT CCGTTA-AGA ATGGTGTGAA CGGAACCCTA GTCGGTAATG GGTTATCTGG
Hp(I) *****T**** ******-*** ********** T********* ********** ********-C
Hp(21) *-*-*--*-* ******-*** *******A** T********* **T******* ****G***--
Hp(9) -----*-*** ******G-AG **A******G G********* ********** ****G***A
310 320 330 340 350 360
Hp(A) AAT-AG-AG--GCCACA--G CTCTCGCCAA AAATTTTTCG GATATCAAGA AAGAATTGAA
, 百拇医药
Hp(I) **GC**-*A--******--A****TT*T** ***C****** **C******* ****G*****
Hp(21) **--**C**AA ******--A* G***A**** ********** **C****G** **********
Hp(9) **C-**-*A--******A A******-*-** ********** **C****G** G*********
370 380 390 400 410 420
Hp(A) TG-AGAA---ATT-TAA--A AATTTCAATA ACAATAACAA TAATGGTCTC AAAAACGGCG
Hp(I) **C*-**--- *C*-*GG--* ********** ***** ***** ******A*** *******---
, 百拇医药
Hp(21) C*-****GTT ***-*GG--* ****C***** *******--- ******A*** ******AA*A
Hp(9) C*-****--- ***A*TTGG* ****C***** *******--- ******A*** ******A***
430 440 450 460 470 480
Hp(A) GAGAACCCAT TTATGCTCAA GTTAATAAAA AGAAAACAGA ACAAGTAGCT AGCCCTGAAG
Hp(I) -*-*-***** *******A** ********** *****G***G G****C**** ****T*****
Hp(21) C***G***** ********** **C******* *********G *****C**** **********
, 百拇医药
Hp(9) C***G**T** ***C****** ********** *****G***G *****C**** **********
490 500 510 520 530 540
Hp(A) AACCCATTTA TGCTCAAGTT GCTAAAAAGG TAACTAAAAA AATTGACCAA CTCAATC-AA
Hp(I) ********** C********* ********** ***A*GC*** ********G* *******-**
Hp(21) *G**T***** C********* ********** *G*G*GC*** ********** *****-*G**
Hp(9) *G******** C********* ********** *G*G*GC*** ********** *****-*G**
, http://www.100md.com
550 560 570 580 590 600
Hp(A) GC-AG--C-G-A---------C-AA---G-----T-G---G--TT-------T--C----
Hp(I) AT-**--*---* -----------*----*-----*-*---*--**-------*--G----
Hp(21) **T*C AT*A*C*---------AT**ATA*A AAAA*T*ACC* GA**AACAA AA*TG*---A
Hp(9) **T*C AT*-A-*A AATAAAT G*-**---*---AA*T*A CC*GA**AA CAA AA*TG*ATCA
610 620 630 640 650 660
, 百拇医药
Hp(A) ---G--G-T---GGTGTAGG-G----CA----AGC-GG-G--C---T-----T---CCC-
Hp(I) ---*--*-*---*T******-*----**----***A*CG*-G*---*-----*---***-
Hp(21) TCA*CA*G*A AA**A**G **C*GTTT**GT GG***A**C*AT*AGC*AGC CC*GAA***A
Hp(9) TCA*--*-*A AA**A**G **C*GTTT**GT GG***A**C*AT*AGC*AGC CC*GAA***A
670 680 690 700 710 720
Hp(A) TTT--G--A-AA---A----G--G---CA--T------G-A---T--------A-A-AG
, 百拇医药
Hp(I) ***--*--*-**---*----*--* ---**--*------*-*---*--------*-*--**
Hp(21) ***AC*CT*C **TTG *TTTT *AT * AAG**--* AATCAA*C *GGC*-TC CCTTT*-*GG**
Hp(9) ***AC*CT*C AA---A----*AT * AAG**AA* --TCAA*C *GGC*-TC CCTTG*G*--**
730 740 750 760 最大同源性
Hp(A) ---T----TG-A---A--GA TCTCAGTAAG GTAGGGCGA-
Hp(I) ---*----**-*----T--** ********** ******---- 87%
, 百拇医药
Hp(21) --A*ACGC** C*GTT*AT** ********** *********- 69% 69%
Hp(9) AAG* -GC** C*GTT*AT** ********** *********- 69% 69% 94%
图2 幽门螺杆菌cagA基因3端片段核苷酸同源性分析. Hp(A):参考文献[4]菌株;Hp(I):参考文献[3]菌株;Hp(21)和Hp(9):临床分离菌株.
*表示相同核苷酸,-表示核苷酸缺失.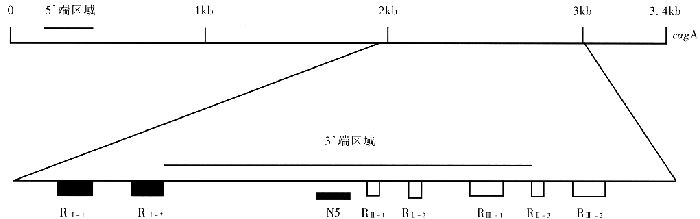
图3 幽门螺杆菌cagA基因重复序列区域结构示意图.
RⅠ:GAATTCAAAAATGGCAAAAATAAGGATTTAAGCAAG RⅡ:GAACCCATTTACGCT
, 百拇医药
RⅢ:TTCCCTTTGAAAAGGCATGATAAAGTTGATGATCTCAGTAAGGTAGGG N5:AATAACAATAACAATAAT
3 讨论
在不同的Hp Ⅰ型菌株之间的cagA基因的大小存在着差异,这主要是因为cagA中一段102bp的DNA重复序列出现的数目不一致造成的. 我们的结果与两个国外发表的Hp cagA基因核苷酸序列[3,4]进行同源性比较,显示Hp cagA 5端片段比较保守,各菌株之间核苷酸序列的最大同源性均在89%以上(图1). 而3端片段核苷酸序列变异比较大,上海地区Hp菌株与欧美菌株之间核苷酸的最大同源性在69%,但是上海地区Hp菌株之间的核苷酸序列的最大同源性为94%~95%. 在cagA基因3端片段核苷酸序列范围内,两株中国Hp菌株比已经报道的两株欧美菌株长近100bp核苷酸. 而欧美菌株之间以及中国菌株之间则仅相差1~2个氨基酸,这说明Hp基因变异有明显的地理区域性. Hp在基因组水平上的变异以及多样性已经有较多的报道并得到比较一致的结果[4,5],但至今仍然没有充分的证据表明cagA基因组水平的变异反反映在蛋白质抗原水平上的差别. 这提示抗原大小的差异很可能是Hp cagA抗原本身所特有的. 变异的原因可能与环境的选择性压力有关.
, 百拇医药
Hp cagA基因的另一个特点是有3个重复序列,它们集中在2kb~3kb范围内,不同的菌株重复序列重复的次数不同,从而造成了CagA抗原大小不一. 美国发表的一个Hp菌株包含了所有3个重复序列,意大利发表的一个Hp菌株则缺失重复序列RⅢ-3和RⅢ-2. 中国Hp菌株cagA 3端片段核苷酸序列分析的范围仅包括重复序列RⅡ-1,RⅡ-2和RⅢ-1. 两株中国Hp菌株均缺失重复序列RⅢ-1. 病原体基因组中的重复序列通常是病原体逃避宿主免疫攻击的一种保护性机制,但是Hp菌株之间的cagA基因重复序列差异形成的机制还不清楚. 文献报道Hp具有转化能力,它提示通过死亡的Hp DNA的转化可以导致基因组的重排,从而导致了基因的变异[3,4]. 日本的研究指出,根据Hp cagA基因重复序列的数目及其排列顺序的不同,可以将cagA基因分为4个基因亚型,不同基因亚型的菌株与不同的胃、十二指肠疾病有关[6]. 采用缺失突变的方法可以更加详细地研究Hp CagA抗原重复序列的确切功能.
, 百拇医药
作者简介:钟平,男,1956-08-22生,上海市人,汉族. 1985年上海电视大学医学系毕业,1991/1994年比利时皇家热带病医学研究所访问研究员. 副研究员,研究室主任,兼任国家药品监督管理局药品审评专家和卫生部艾滋病专家咨询委员会委员,主要从事临床诊断试剂的研制和艾滋病研究工作,国内外刊物发表论文7篇.
通讯作者 钟平,200052,上海市延安西路1262号,上海生物制品研究所诊断试剂研究室.
Correspondence to:ZHONG Ping, Laboratory of Diagnostic Reagents, Shanghai Institute of Biological Products, 1262 Yanan Xilu, Shanghai 200052, China
Tel. +86.21.62803189 Ext.147, Fax.+86.21.62801807
, 百拇医药
Email. pzhongc@online.sh.cn
4 参考文献
1 Labigne A, Cussac V, Courcoux P. Shuttle cloning and nucleotide sequences of Helicobacter pylori genes responsible for urease activity. J Bacteriol, 1991;173:1920-1931
2 分子克隆实验指南. J.萨姆布鲁克,E.F.弗里奇,T. 曼尼阿蒂斯著. 金冬雁,黎孟枫译. 北京科学出版社,1992年10月第二版.
3 Covacci A, Censini S, Bugnoli M, Petracca R, Burroni D, Macchia G, Massone A, Papini E, Xiang ZY, Figura N, Pappuoli R. Molecular characterization of the 128-kDa immunodominant antigen of Helicobacter pylori associated with cytotoxicity and duodenal ulcer. Proc Natl Acad Sci USA, 1993;90:5791-5795
, 百拇医药
4 Tummuru MKR, Cover TL, Blaser MJ. Cloning and expression of a high-molecular-mass major antigen of Helicobacter pylori:evidence of linkage to cytotoxin production. Infect Immun, 1993;61:1799-1809
5 Akopyanz N, Bukanov NO, Westblom TU, Kresovich S, Berg DE. DNA diversity among clinical isolates of Helicobacter pylori detected by PCR-based RAPD fingerprinting. Nucleic Acids Res, 1992;20:5137-5142
6 Yamaoka Y, Kodama T, Kashima K, Graham DY, Sepulveda AR. Variants of the 3 region of the cagA gene in Helicobacter pylori isolates from patients with different H. pylori-associated diseases. J Clin Microbiol, 1998;36:2258-2263
收稿日期 1999-04-08 修回日期 1999-06-03, http://www.100md.com
单位:钟 平 吴昆详 徐帆洪 上海生物制品研究所诊断试剂研究室 上海市 200052; 许春娣 徐家裕 上海瑞金医院小儿内科 上海市 200025
关键词:幽门螺杆菌感染;细胞毒素相关基因A;基因克隆;cagA序列分析
世界华人消化杂志990810 中国图书馆分类号 R573
摘 要
目的 了解和分析中国幽门螺杆菌(Helicobacter pylori, Hp)菌株细胞毒素相关基因A(cagA)的核苷酸序列特性.
方法 从两位患胃溃疡儿童的胃粘膜中分离两株Hp菌株,用PCR方法扩增cagA基因的5端和3端的两个片段并作克隆和序列分析.
, http://www.100md.com
结果 两株中国Hp菌株cagA基因之间的同源性为94%~95%. 中国Hp菌株与国外菌株相同区段比较,5端的同源性为89%~92%,3端的同源性为69%.
结论 中国Hp菌株与国外Hp菌株cagA基因5端序列部分区域的同源性较好,可以作为分子检测Hp感染的靶基因序列,而cagA基因3端序列部分区域的同源性较差,可以作为Hp分子流行病学研究的靶基因序列.
Cloning and characterization of Hp cagA gene sequences derived from Chinese strains
ZHONG Ping1, WU Kun-Xiang1, XU Fan-Hong1, XU Chun-Di2 and XU Jia-Yu2
, 百拇医药
1Laboratory of Diagnostic Reagents, Shanghai Institute of Biological Products, Shanghai 200052, China
2Shanghai Ruijin Hospital, Shanghai 200025, China
Subject headings Helicobacter pylori infection; cagA; gene cloning; cagA gene; sequencing
Abstract
AIM To investigate the sequence characterization of Hp cagA gene from Chinese strains.
, 百拇医药
METHODS Two Hp strains derived from two Chinese pediatric patients with gastric ulcer were isolated. Two DNA fragments of Hp cagA gene 5 and 3 regions of each strain were PCR-amplified, cloned and sequenced.
RESULTS The nucleotide homology between two Chinese Hp strains was 94%. However, the nucleotide homology of cagA gene 5 and 3 regions between Chinese and American strains and between Chinese and Italian strains were 89% and 69%-71%, respectively.
, 百拇医药
CONCLUSION The nucleotide sequences of Hp cagA gene 5 region among the strains showed a better homology, this region could be chosen as a target gene for molecular diagnosis of Hp infection, while that of cagA gene 3 region between Chinese and American strains or between Chinese and Italian strains showed a poorer homology, this region could be chosen as a target gene for Hp molecular epidemiological study.
0 引言
幽门螺杆菌(Helicobacter pylori, Hp)是消化性溃疡和慢性胃炎的主要致病因子,并与胃癌的发生有密切关系. 目前国外已经发表了近10个Hp菌株cagA基因的全序列,然而中国Hp菌株的cagA基因的序列还未见报道. 为此,我们从中国患胃溃疡儿童胃粘膜中分离了两株Hp,进行分子克隆和分析了它们的cagA基因中的两个片段的核苷酸序列,为中国分子诊断Hp感染和进行Hp的分子流行病学研究奠定基础.
, 百拇医药
1 材料和方法
1.1 材料 胃粘膜组织取自于上海第二医科大学瑞金医院儿科两位胃溃疡儿童. Hp分离培养基:培养基成分为:Campylobacter Agar Base 43g/L(OXOID),奈啶酸20mg/L(Sigma),TTC 40mg/L (F.M.K.),万古霉素6mg/L(Sigma),两性霉素B2mg/L(Sigma),100mL/L新鲜羊血. pMAL-C2购自于美国Biolabs公司,pSP72和大肠杆菌JM109由本所分子生物学研究室提供. 限制性内切酶和T4 DNA连接酶(Boehringer Mannheim), Taq plusⅡ DNA聚合酶(上海Sangon公司),T7测序试剂盒(Pharmacia),α-35S-dCTP(Amersham). Hp cagA 5端核苷酸序列片段的PCR扩增引物(cagA基因157nt~504nt):P1(上游引物):5-CCGGAGAATTCGATAACAGGCAAGCTTTTGAGG-3;P2(下游引物):5-GCCTGCAGTAATCGAAAAGA_TTGTTTGGCAG-3;Hp cagA 3端核苷酸序列片段的PCR扩增引物(cagA基因2269nt~2877nt):P3(上游引物):5-GCGGATCCAGCAAGGTAACGCAAGC-3;P4(下游引物):5-GCGTCGACTTATCGCCCTACTTCACTGA_GATC-3. 引物由上海Sangon公司合成.
, 百拇医药
1.2 方法 内镜下应用常规方法在胃窦部炎症明显处铗取胃粘膜组织块,接种于选择培养基平板,置37℃微氧环境(50mL/L O2,70mL/L CO2,80mL/L H2和800mL/L N2)下培养5d. Hp基因组DNA的制备参照文献[1]中的方法. PCR扩增cagA基因5端和3端片段:PCR反应体积为50μL,其中包括主要试剂0.3μmol/L引物,1.5mmol/L Mg2+,0.5U Taq DNA聚合酶. PCR反应条件:94℃ 4min预变性;94℃ 1min,55℃ 1min,72℃ 1min,共35个循环;72℃延伸10min. DNA片段纯化、质粒连接和细菌转化参照文献[2]方法进行. DNA序列测定和分析采用Sanger双脱氧链终止法测序. 采用电脑DNA分析辅助软件对所测得的DNA序列进行同源性分析.
2 结果
, http://www.100md.com
2.1 PCR扩增cagA基因片段 5端片段PCR片段扩增产物长度为340bp,与预期的核苷酸长度相符合;3端片段PCR扩增产物的长度为700bp,比预期的长度604bp长.
2.2 测序质粒的构建 PCR扩增的Hp cagA基因5端片段和3端片段分别经EcoRⅠ, PstⅠ和BamHⅠ, SalⅠ双酶切反应后分别定向插入于载体pMAL-C2和pSP72中,得到四个测序重组质粒如pMA1, pMA2, pSB1和pSB2. 它们的双酶切反应后的片段与预期的核苷酸长度结果相符合.
2.3 Hp cagA基因5端和3端部分核苷酸序列和同源性分析 Hp cagA基因5端部分核苷酸序列中,两株中国Hp菌株(SH21和SH9)之间的最大同源性为94%~95%,它们与美国和意大利Hp菌株核苷酸之间的最大同源性为89%~92%(图1). Hp cagA基因3端部分核苷酸序列中,两株中国Hp菌株(SH21和SH9)之间的最大同源性为94%,而它们与美国和意大利Hp菌株核苷酸之间的最大同源性为69%(图2).
, http://www.100md.com
10 20 30 40 50 60
Hp(A) GATAACAGGC AAGCTTTTGA GGGAATCTCG CAATAAAGGG AAGAATACTC CAATAAAGCG
Hp(I) ********** ********** A********* ****T***** ********** **********
Hp(21) ********** ********** **A******* ***CT***** ********G* **********
Hp(9) ********** ********** **A******* ***CT***** ********G* **********
70 80 90 100 110 120
, 百拇医药
Hp(A) ATCAAAAATC CTACCAAAAA GAATCAGTAT TTTTCAGACT TTATCAATAA GAGCAATGAT
Hp(I) ********** ********** ********** ********** ********** **********
Hp(21) ********** *C******** ********** ********** ********** **********
Hp(9) ********** *CG******* ********** ********** ********** **********
130 140 150 160 170 180
Hp(A) TTAATCAAGA AAGACAATCT CATTGTCGTG GAATCTTCCA CAAAGAGCTT TCAGAAATTT
, http://www.100md.com
Hp(I) ********C* ********** *****AT**A ********** ********** **********
Hp(21) **G*****C* ********** *****CT**A **T******G T*G******* *A********
Hp(9) **G*****C* ********** *****CT**A **T******G T*G*T***** *A********
190 200 210 220 230 240
Hp(A) GGGGATCAGC GTTACCGAAT TTTCACAAGT TGGGTGTCCC ATCAAAACGA TCCGTCTAAA
Hp(I) ********** ********** ********** ********** ********** **********
, 百拇医药
Hp(21) ********** ******A*** ***T**G*** ********** T******A** **********
Hp(9) ********** ******A*** ***T**G*** ********** T******A** **********
250 260 270 280 290 最大同源性
Hp(A) ATCAACACCC GATGCATCCG AAATTTTATG GAACATACCA TACAACCCCC TATCCCT
Hp(I) ********** ***CG***** ********** ***A***T** ********** *****T* 96%
Hp(21) ********** A*CAA***** ********** ***A***TT* ********** ****T** 90%92%
, 百拇医药
Hp(9) ********** A*CAA****A ********** ***A***T** ********** ****T** 89%90%95%
图1 幽门螺杆菌cagA基因5端片段核苷酸同源性分析. Hp(A):参考文献[4]菌株;Hp(I):参考文献[3]菌株;Hp(21)和Hp(9):临床分离菌株.
*表示相同核苷酸.
10 20 30 40 50 60
Hp(A) TTCAGCAAGG TAACGCAAGC AAAAAGCGAC CAAGAAAATT CCATTAAAGA TGTGATCATC
Hp(I) ********** ********** ********** *TT******* **G******* **********
, 百拇医药
Hp(21) ********** ********** ********** *TT******* *******G** *********T
Hp(9) ********** ********** ********** *TT******* *******G** **********
70 80 90 100 110 120
Hp(A) AATCAAAAGA TAACGGATAA AGTTGATGAA-CTCAATCAA GCGGTATCAG TGGCTAAAAT
Hp(I) *********G ********** *******A*T-********* ********** ********GC
Hp(21) ********** ********** ******C-** T******** G **T******* A*A*****T*
, 百拇医药
Hp(9) ********** ********** ******A-** T******** G **T**G**** A*A*****T*
130 140 150 160 170 180
Hp(A) AGC-GTGCGA TTTCAGTGGG GTAGAGCAAG CGTTAGCCGA TCTCAAAAAT TTCTCAAAGG
Hp(I) *A*-*G*T** *******A** ********** *CC******* ********** **********
Hp(21) A*A-G***** *******AA* ********** CC******** *********C **G***TT**
Hp(9) *A*A*-**** *******AA* ********** *CC******* A******GC* **G***TT**
, 百拇医药
190 200 210 220 230 240
Hp(A) AGCAATTGGC TCAACAAGCT CAAAAAAATG AAAGTT-TCA ATGTTGGA- - - - - - TCTGA
Hp(I) ********** C********* ********** **-**CC*** ***C*A**AA AAAA******
Hp(21) *T*--****---**-**----**-----*-----*---* -**----*------***CC*
Hp(9) *T*--****---**-**----**-----*-----*---* -**----*------***-*-
250 260 270 280 290 300
, 百拇医药
Hp(A) ATATACCAAT CCGTTA-AGA ATGGTGTGAA CGGAACCCTA GTCGGTAATG GGTTATCTGG
Hp(I) *****T**** ******-*** ********** T********* ********** ********-C
Hp(21) *-*-*--*-* ******-*** *******A** T********* **T******* ****G***--
Hp(9) -----*-*** ******G-AG **A******G G********* ********** ****G***A
310 320 330 340 350 360
Hp(A) AAT-AG-AG--GCCACA--G CTCTCGCCAA AAATTTTTCG GATATCAAGA AAGAATTGAA
, 百拇医药
Hp(I) **GC**-*A--******--A****TT*T** ***C****** **C******* ****G*****
Hp(21) **--**C**AA ******--A* G***A**** ********** **C****G** **********
Hp(9) **C-**-*A--******A A******-*-** ********** **C****G** G*********
370 380 390 400 410 420
Hp(A) TG-AGAA---ATT-TAA--A AATTTCAATA ACAATAACAA TAATGGTCTC AAAAACGGCG
Hp(I) **C*-**--- *C*-*GG--* ********** ***** ***** ******A*** *******---
, 百拇医药
Hp(21) C*-****GTT ***-*GG--* ****C***** *******--- ******A*** ******AA*A
Hp(9) C*-****--- ***A*TTGG* ****C***** *******--- ******A*** ******A***
430 440 450 460 470 480
Hp(A) GAGAACCCAT TTATGCTCAA GTTAATAAAA AGAAAACAGA ACAAGTAGCT AGCCCTGAAG
Hp(I) -*-*-***** *******A** ********** *****G***G G****C**** ****T*****
Hp(21) C***G***** ********** **C******* *********G *****C**** **********
, 百拇医药
Hp(9) C***G**T** ***C****** ********** *****G***G *****C**** **********
490 500 510 520 530 540
Hp(A) AACCCATTTA TGCTCAAGTT GCTAAAAAGG TAACTAAAAA AATTGACCAA CTCAATC-AA
Hp(I) ********** C********* ********** ***A*GC*** ********G* *******-**
Hp(21) *G**T***** C********* ********** *G*G*GC*** ********** *****-*G**
Hp(9) *G******** C********* ********** *G*G*GC*** ********** *****-*G**
, http://www.100md.com
550 560 570 580 590 600
Hp(A) GC-AG--C-G-A---------C-AA---G-----T-G---G--TT-------T--C----
Hp(I) AT-**--*---* -----------*----*-----*-*---*--**-------*--G----
Hp(21) **T*C AT*A*C*---------AT**ATA*A AAAA*T*ACC* GA**AACAA AA*TG*---A
Hp(9) **T*C AT*-A-*A AATAAAT G*-**---*---AA*T*A CC*GA**AA CAA AA*TG*ATCA
610 620 630 640 650 660
, 百拇医药
Hp(A) ---G--G-T---GGTGTAGG-G----CA----AGC-GG-G--C---T-----T---CCC-
Hp(I) ---*--*-*---*T******-*----**----***A*CG*-G*---*-----*---***-
Hp(21) TCA*CA*G*A AA**A**G **C*GTTT**GT GG***A**C*AT*AGC*AGC CC*GAA***A
Hp(9) TCA*--*-*A AA**A**G **C*GTTT**GT GG***A**C*AT*AGC*AGC CC*GAA***A
670 680 690 700 710 720
Hp(A) TTT--G--A-AA---A----G--G---CA--T------G-A---T--------A-A-AG
, 百拇医药
Hp(I) ***--*--*-**---*----*--* ---**--*------*-*---*--------*-*--**
Hp(21) ***AC*CT*C **TTG *TTTT *AT * AAG**--* AATCAA*C *GGC*-TC CCTTT*-*GG**
Hp(9) ***AC*CT*C AA---A----*AT * AAG**AA* --TCAA*C *GGC*-TC CCTTG*G*--**
730 740 750 760 最大同源性
Hp(A) ---T----TG-A---A--GA TCTCAGTAAG GTAGGGCGA-
Hp(I) ---*----**-*----T--** ********** ******---- 87%
, 百拇医药
Hp(21) --A*ACGC** C*GTT*AT** ********** *********- 69% 69%
Hp(9) AAG* -GC** C*GTT*AT** ********** *********- 69% 69% 94%
图2 幽门螺杆菌cagA基因3端片段核苷酸同源性分析. Hp(A):参考文献[4]菌株;Hp(I):参考文献[3]菌株;Hp(21)和Hp(9):临床分离菌株.
*表示相同核苷酸,-表示核苷酸缺失.

图3 幽门螺杆菌cagA基因重复序列区域结构示意图.
RⅠ:GAATTCAAAAATGGCAAAAATAAGGATTTAAGCAAG RⅡ:GAACCCATTTACGCT
, 百拇医药
RⅢ:TTCCCTTTGAAAAGGCATGATAAAGTTGATGATCTCAGTAAGGTAGGG N5:AATAACAATAACAATAAT
3 讨论
在不同的Hp Ⅰ型菌株之间的cagA基因的大小存在着差异,这主要是因为cagA中一段102bp的DNA重复序列出现的数目不一致造成的. 我们的结果与两个国外发表的Hp cagA基因核苷酸序列[3,4]进行同源性比较,显示Hp cagA 5端片段比较保守,各菌株之间核苷酸序列的最大同源性均在89%以上(图1). 而3端片段核苷酸序列变异比较大,上海地区Hp菌株与欧美菌株之间核苷酸的最大同源性在69%,但是上海地区Hp菌株之间的核苷酸序列的最大同源性为94%~95%. 在cagA基因3端片段核苷酸序列范围内,两株中国Hp菌株比已经报道的两株欧美菌株长近100bp核苷酸. 而欧美菌株之间以及中国菌株之间则仅相差1~2个氨基酸,这说明Hp基因变异有明显的地理区域性. Hp在基因组水平上的变异以及多样性已经有较多的报道并得到比较一致的结果[4,5],但至今仍然没有充分的证据表明cagA基因组水平的变异反反映在蛋白质抗原水平上的差别. 这提示抗原大小的差异很可能是Hp cagA抗原本身所特有的. 变异的原因可能与环境的选择性压力有关.
, 百拇医药
Hp cagA基因的另一个特点是有3个重复序列,它们集中在2kb~3kb范围内,不同的菌株重复序列重复的次数不同,从而造成了CagA抗原大小不一. 美国发表的一个Hp菌株包含了所有3个重复序列,意大利发表的一个Hp菌株则缺失重复序列RⅢ-3和RⅢ-2. 中国Hp菌株cagA 3端片段核苷酸序列分析的范围仅包括重复序列RⅡ-1,RⅡ-2和RⅢ-1. 两株中国Hp菌株均缺失重复序列RⅢ-1. 病原体基因组中的重复序列通常是病原体逃避宿主免疫攻击的一种保护性机制,但是Hp菌株之间的cagA基因重复序列差异形成的机制还不清楚. 文献报道Hp具有转化能力,它提示通过死亡的Hp DNA的转化可以导致基因组的重排,从而导致了基因的变异[3,4]. 日本的研究指出,根据Hp cagA基因重复序列的数目及其排列顺序的不同,可以将cagA基因分为4个基因亚型,不同基因亚型的菌株与不同的胃、十二指肠疾病有关[6]. 采用缺失突变的方法可以更加详细地研究Hp CagA抗原重复序列的确切功能.
, 百拇医药
作者简介:钟平,男,1956-08-22生,上海市人,汉族. 1985年上海电视大学医学系毕业,1991/1994年比利时皇家热带病医学研究所访问研究员. 副研究员,研究室主任,兼任国家药品监督管理局药品审评专家和卫生部艾滋病专家咨询委员会委员,主要从事临床诊断试剂的研制和艾滋病研究工作,国内外刊物发表论文7篇.
通讯作者 钟平,200052,上海市延安西路1262号,上海生物制品研究所诊断试剂研究室.
Correspondence to:ZHONG Ping, Laboratory of Diagnostic Reagents, Shanghai Institute of Biological Products, 1262 Yanan Xilu, Shanghai 200052, China
Tel. +86.21.62803189 Ext.147, Fax.+86.21.62801807
, 百拇医药
Email. pzhongc@online.sh.cn
4 参考文献
1 Labigne A, Cussac V, Courcoux P. Shuttle cloning and nucleotide sequences of Helicobacter pylori genes responsible for urease activity. J Bacteriol, 1991;173:1920-1931
2 分子克隆实验指南. J.萨姆布鲁克,E.F.弗里奇,T. 曼尼阿蒂斯著. 金冬雁,黎孟枫译. 北京科学出版社,1992年10月第二版.
3 Covacci A, Censini S, Bugnoli M, Petracca R, Burroni D, Macchia G, Massone A, Papini E, Xiang ZY, Figura N, Pappuoli R. Molecular characterization of the 128-kDa immunodominant antigen of Helicobacter pylori associated with cytotoxicity and duodenal ulcer. Proc Natl Acad Sci USA, 1993;90:5791-5795
, 百拇医药
4 Tummuru MKR, Cover TL, Blaser MJ. Cloning and expression of a high-molecular-mass major antigen of Helicobacter pylori:evidence of linkage to cytotoxin production. Infect Immun, 1993;61:1799-1809
5 Akopyanz N, Bukanov NO, Westblom TU, Kresovich S, Berg DE. DNA diversity among clinical isolates of Helicobacter pylori detected by PCR-based RAPD fingerprinting. Nucleic Acids Res, 1992;20:5137-5142
6 Yamaoka Y, Kodama T, Kashima K, Graham DY, Sepulveda AR. Variants of the 3 region of the cagA gene in Helicobacter pylori isolates from patients with different H. pylori-associated diseases. J Clin Microbiol, 1998;36:2258-2263
收稿日期 1999-04-08 修回日期 1999-06-03, http://www.100md.com