中国大麦黄花叶病毒分离物的分子变异
作者:施农农 陈剑平 M.J.Adams
单位:施农农 陈剑平 浙江省农业科学院,浙江 杭州 310021;M.J.Adams 英国洛桑试验站
关键词:大麦黄花叶病毒;分子变异;单链构象多态性;序列分析;限制性切酶图谱
病毒学报990407 摘要:13个供试的中国和英国大麦黄花叶病毒(BaYMV)分离物,经RNA1和RNA2全基因组不同区域DNA片段单链构象多态性分析(SSCP),外壳蛋白基因和RNA2 70kD基因5'端705碱基序列分析,以及此705碱基DNA片段限制性内切酶图谱分析结果,它们的RNA1和RNA2彼此无一相同,其中RNA2变异比RNA1更大。由于变异十分复杂,且没有规律性,因而当前通用的分子生物学技术尚不能简单地用于BaYMV致病性分化或株系区分和检测。
中图分类号:S435.121.4 文献标识码:A 文章编号:1000-8721(1999)04-0339-09
, http://www.100md.com
MOLECULAR VARIATIONS OF CHINESE ISOLATES OF BARLEY YELLOW MOSAIC BYMOVIRUS
SHI Nong-nong1, CHEN Jian-ping1, M. J. Adams2
(1. Zhejiang Academy of Agricultural Sciences, Zhejiang,Hangzhou 310021, China;
2.IACR-Rothamsted, Harpenden, Herts, UK AL5 2JQ)
Abstract: DNA fragments amplified from the bipartite genome of thirteen Chinese and UK isolates of barley yellow mosaic bymovirus(BaYMV) by RT-PCR were analysed by single-strand conformation polymorphism(SSCP). Variations in the coat protein gene and the 5'-terminal part(c. 705bp) of the 70kD regions of RNA2 of these BaYMV isolates were also revealed by sequencing and restriction mapping. The results indicated that none of all thirteen isolates was identical each other at the molecular level. Differences in RNA2 were greater than those in RNA1. The variations were very complicated so that none of modern molecular techniques could simply correlate the variations at nucleotide level with pathogenicity or strain differentiation.
, 百拇医药
Key words: Barley yellow mosaic bymovirus; Molecular variation; Single-strand conformation polymorphism; Sequencing ;Restriction mapping
大麦黄花叶病毒(Barley yellow mosaic virus, BaYMV)是日本、中国和西北欧冬大麦的一种重要病害,是马铃薯Y病毒科大麦黄花叶病毒属(Bymovirus)的代表种,含有2个单链正链RNA基因组(RNA1为7.6kb,RNA2为3.5kb)。日本和德国BaYMV核酸全序列已测定[1-4],二者RNA1和RNA2的同源性分别为93.6%和90.4%。
BaYMV由土壤中的禾谷多粘菌Polymyxa graminis传播,轮作和农药无明显防病作用,唯一有效的防治措施是栽培抗病品种。然而,不同国家的BaYMV在致病性方面存在差异。已经表明,日本的BaYMV存在6个株系[5],欧洲的至少存在2个株系[6],我国也存在6个株系,且与日本和欧洲的株系都不同[7],已经用单链构象多态性-聚合酶链反应初步研究了不同BaYMV的变异情况[8,9]。本文应用反转录-聚合酶链反应(RT-PCR)、单链构象多态性分析(SSCP)、核酸序列分析和限制性内切酶图谱分析等分子生物学技术,进一步研究了我国BaYMV分离物的分子变异。
, http://www.100md.com
材料与方法
1 病毒分离物 1994年3月中旬和1995年3月上旬,分别从江苏省阜宁县施庄和盐城市郊区、扬州里下河地区农科所农场、如东县农科所农场、海安县农科所农场、南通市农科所农场、浙江省嘉善县良种场、浙江省农科院农场、宁波市妙山农场等地BaYMV病田,以及江苏沿海地区农科所上海病圃,采集具有典型BaYMV症状的大麦叶片,-70℃贮藏或冷冻干燥后备用。英国3个BaYMV分离物[9]由M.J.Adams提供,其中UK-1为欧洲普通株(BaYMV-1),UK-2和UK-3为抗性克服株(BaYMV-2)。
2 植物总RNA抽提 抽提方法参见文献[10]。
3 RT-PCR 分别用与BaYMV RNA1或RNA2 3'端互补的引物N11或N8(表1),以RNA1或RNA2为模板合成第一链cDNA。10μl植物总RNA,加1μl引物(500ng/μl),70℃加热10分钟,迅速在冰中冷却;然后加4μl 5×H-RT缓冲液(Life Technologies Ltd),2μl 0.1mol/L DTT,1μl 10mmol/L dNTPs和1μl水,37℃水浴2分钟;最后加1μl(200U)逆转录酶(Life Technologies Ltd),37℃水浴1小时。合成完成后,加水至总体积100μl,-20℃贮藏备用。
, 百拇医药
表1 用于BaYMV基因组扩增的引物
Table 1 Oligonucleotide primers used to amplify parts of the BaYMV genome 引物a
Primer
核苷酸位置b
Nucleotide position
序列
Sequence
温度
Tm(℃)d
, http://www.100md.com RNA1
N1(+)
30-47
5'-CAAAACGAAACAAACACA-3'
43
N26(+)
601-620
5'-CTGCAAGAGAGCTCTTCTTC-3'
55
N27(+)
1204-1223
5'-AGCGTCGTTGGGATCACTTA-3'
, http://www.100md.com
55
N28(+)
1801-1820
5'-CATGAGGGTTGGTCCAGAAT-3'
55
N29(+)
2412-2431
5'-ATTGCGTCTTGATCGACGCA-3'
55
N30(+)
3011-3030
5'-CACGCGTTGTCACTCTACAA-3'
, http://www.100md.com
55
N15(+)
3620-3638
5'-ATATGGATGCAGCCGTTGA-3'
51
N22(+)
4848-4867c
5'-TGATAGACCGGCTTGTGGGT-3'
57
N37(+)
5778-5797
, 百拇医药
5'-CTCCATCGATCCTCTCTTCT-3'
55
N12(+)
6508-6524
5'-CAAGCTGCTGATCCTCT-3'
47
N14(+)
6760-6776
5'-GATGGTAACTGGAGCGT-3'
47
N31(-)
, http://www.100md.com
800-781
5'-TTTAAGCCGCGTAGCATAGC-3'
55
N32(-)
1387-1368
5'-TCGTTGTGACTGCATCCTCA-3'
55
N33(-)
2020-2001
5'-CATCTCTGGGAGTTGCTCAA-3'
55
, 百拇医药
N34(-)
2575-2556
5'-TAAAACTCAGGAGAGCGGCA-3'
55
N35(-)
3211-3192
5'-CCGTGCATCTTCGCAAGTTT-3'
55
N36(-)
3813-3794
5'-GGTCACCTTCTTACGCCTTT-3'
, http://www.100md.com
55
N17(-)
4970-4952
5'-CCCTCAATGGCATTGTTGA-3'
51
N23(-)
6119-6100c
5'-TCGTGCCCAAACTCTTCTTC-3'
55
N16(-)
6578-6561
, 百拇医药
5'-AAGCGTGCTCCATCAGCT-3'
51
N13(-)
7407-7391
5'-GGTTTAGGTTAGTTCTG-3'
43
N11(-)
7632-7616
5'-ATTACCTTCTGGTACTC-3'
43
RNA2
, 百拇医药
N1(+)
30-47
5'-CAAAACGAAACAAACACA-3'
43
N9(+)
943-959
5'-CACAAATTGGAACTCTG-3'
43
N3(+)
1520-1536
5'-GAACCAGTTGCTAGTGG-3'
, 百拇医药
47
N4(+)
2609-2625
5'-CAAGCATCCAATCTCAG-3'
45
N5(-)
1281-1265
5'-AAACCGCCAAGATTGAT-3'
43
N10(-)
1647-1631
5'-TGGAATTTCACACGCTT-3'
, 百拇医药
43
N6(-)
1853-1837
5'-AGACAACCTCCCCAAAA-3'
45
N7(-)
2948-2932c
5'-ACCGCTAACTCTACCAT-3'
45
N8(-)
3585-3569
, 百拇医药 5'-GTCACATTTCCTGTGTA-3'
43
a.(+)病毒RNA序列,(-)病毒RNA互补序列;b.引物根据日本BaYMV序列设计(Kashiwazaki, et al.1990,1991);c.根据德国BaYMV序列设计,与日本序列有1-2个碱基差异;d.根据经验公式(A+T)×2+(C+G)×4-5计算。
a.(+)Matching, (-)Complementary to virus sequence; b. Primers designed according to sequences of Japanese BaYMV isolate (Kashiwazaki, et al. 1990, 1991); c. Designed according to the German sequence with 1 or 2 mismatches to the Japanese isolate; d. Calculated as (A+T)×2+(C+G)×4-5.
, http://www.100md.com
用表1、图1所列的引物,以cDNA1和cDNA2为模板,扩增BaYMV RNA1和RNA2 5'端至3'端各区域DNA片段。引物根据已发表的BaYMV核苷酸序列设计。50μl体积PCR反应含1μlcDNA模板,20pmol引物对,200mmol/L dNTPs,1.5mmol/L MgCl2,50mmol/L KCl,10mmol/L Tris-HCl pH8.3和2.5μl Taq DNA聚合酶(GIBCO)。经94℃变性3分钟,进行30个循环扩增,每个循环包括94℃变性1分钟,43℃~55℃退火1分钟,72℃合成1~2分钟。PCR产物经1%或1.5%琼脂糖电泳检测。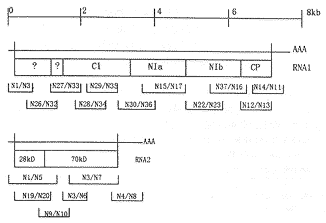
图1 BaYMV RNA1和 RNA2基因组结构及表1中所列进行RT-PCR扩增的引物
Figure 1 Genome organizations of BaYMV RNA1 and RNA2 and DNA fragments amplified by RT-PCR using the primers detailed in Table l
, 百拇医药
4 SSCP分析 参见文献[8,9]。
5 DNA克隆和序列分析 用pCRⅡ(InVitrogen公司产品)和pT7Blue(Novagen公司产品)质粒克隆PCR产物,方法见公司产品说明书。用Sequenase(Version 2.0)DNA序列分析试剂盒(USB)测定PCR克隆的核苷酸序列,方法见试剂盒说明书。
6 DNA酶切图谱分析 根据序列推测酶切位点,选择有关限制性内切酶进行酶切,在一个10μl的酶切反应中,含2μl DNA,1μl相应10×缓冲液,0.5μl酶,37℃水浴45分钟,经1%琼脂糖胶进行酶切图谱分析。有必要时,进一步用Southern blot分析[10]。
结果
1 应用RT-PCR区分不同的BaYMV分离物
, 百拇医药
以10个中国BaYMV分离物和3个英国BaYMV分离物的RNA1和RNA2的cDNA为模板,从5'端至3'端依次用不同引物对扩增不同长度的PCR片段,PCR片段大小分别为334bp至1 429bp,其中RNA1扩增区域为第31~7 632碱基,RNA2为第92~3 585碱基,均接近全长(图1)。
RNA1:用6对引物即N1/N31、N26/N32、N27/N33、N30/N36、N22/N23和N12/N13,从所有10个中国BaYMV RNA1 cDNA模板上都能扩增到预期的DNA片段,但N29/N35则不能扩增。N15/N17能从阜宁、海安、杭州、嘉善、宁波、南通、上海、盐城和扬州分离物上扩增出预期的1 352bp片段,但不能从如东分离物扩增。N14/N11除不能从嘉善分离物扩增外,从其他9个中国分离物上均能扩增到预期的873bp片段。
用引物对N30/N36扩增,所有10个中国分离物均产生一个均匀的PCR片段,但英国分离物UK-1和UK-3仅产生非常微弱的同一PCR片段,大小为803bp,与期望的片段一致,而UK-2则不产生任何产物。此PCR反应重复了4次,结果均相同。用引物对N28/N34扩增,所有3个英国分离物和阜宁、上海分离物均产生一个预期的775bp片段,而其它8个中国分离物均不扩增。用引物对N37/N16扩增后,盐城、宁波、扬州、南通、杭州、嘉善、上海分离物和所有3个英国分离物均扩增一个预期的801bp片段,而阜宁、南通和如东不产生任何产物(表2)。
, http://www.100md.com
RNA2:用6对引物即N1/N5、N19/N20、N9/N10、N3/N6、N3/N7和N4/N8,从10个供试中国BaYMV分离物RNA2模板上,均分别特异地扩增到1 252bp、922bp、705bp、334bp、1 429bp和977bp片段。经电泳分析,根据这些片段,不能区别不同的BaYMV分离物(表2)。
表2 BaYMV RNA1和RNA2 RT-PCR产物单链构象多态性分析(SSCP)图谱综合
Table 2 Summary of patterns of RT-PCR products amplified from BaYMV RNA1 and RNA2 by single-strand conformation polymorphism analysis 分离物
Isolates
RNA1
, 百拇医药
N1/N31a
N26/N32
N27/N33
N28/N34
N29/N35
N30/N36
N15/N17
N22/N23
N37/N16
N12/N13
N14/N11
771bp
, 百拇医药
687bp
817bp
775bp
800bp
803bp
1351bp
1272bp
801bp
900bp
873bp
FU
Ab
A
, 百拇医药
A
A
-c
A
A
A
-
A
A
HA
B
B
A
-
, 百拇医药
-
B
A
B
A
B
HZ
C
C
B
-
-
C
B
, 百拇医药
C
B
C
C
JI
D
A
C
-
-
B
C
A
C
, 百拇医药
D
-
NA
D
D
A
-
-
B
D
A
-
B
A
, 百拇医药
NI
E
E
A
-
-
D
A
A
D
E
B
RU
D
, 百拇医药
C
A
-
-
E
-
A
-
F
A
SH
C
A
B
, 百拇医药
B
-
F
E
A
C
G
D
YC
F
C
A
-
-
, 百拇医药
G
F
D
E
B
E
YZ
D
C
D
-
-
F
F
, 百拇医药
B
F
D
A
分离物
Isolates
RNA2
N1/N5
N19/N20
N9/N10
N3/N7
N3/N6
N4/N8
, http://www.100md.com
1252bp
922bp
705bp
1429bp
334bp
977bp
FU
A
A
A
A
A
A
, 百拇医药
HA
B
B
B
B
A
B
HZ
C
C
C
C
B
C
, http://www.100md.com
JI
D
D
D
D
C
D
NA
E
E
E
E
D
E
, http://www.100md.com
NI
F
F
B
F
E
F
RU
G
G
F
G
A
G
, http://www.100md.com
SH
H
H
G
H
F
H
YC
I
I
B
I
G
I
, http://www.100md.com
YZ
J
D
H
J
H
J
FU:阜宁Funing,HA:海安Haian,HZ:杭州Hangzhou,JI:加善Jiashan,NI:宁波Ningbo, NA:南通Nantong,RU:如东Rudong,SH:上海Shanghai,YC:盐城Yancheng,YZ:扬州Yangzhou。
a.用引物对N1/N31从RNA1扩增的DNA片段(771bp);b.同一列中相同字母表示相同SSCP图谱;c.RT-PCR扩增无产物。
, 百拇医药
a.A fragment amplified from RNA1 template with primer pair N1/N31;b.Same letter means identical SSCP pattern in one column;c.No fragment amplified by RT-PCR.
2 应用SSCP分析区分不同的BaYMV分离物
我们通过SSCP分析,鉴定了中国BaYMV分离物之间的分子差异,表2是SSCP分析结果的综合归纳。
BaYMV基因组变异比较结果表明,10个中国分离物RNA2的变异程度均比RNA1大。所有10个中国分离物N1/N5片段、N3/N7片段和N4/N8片段的SSCP图谱彼此都不相同。就SSCP图谱,N9/N10片段的,只有海安、宁波和盐城分离物相同;N3/N6片段的,只有海安、阜宁和如东分离物相同;N19/N20片段的,只有嘉善与扬州分离物相同。其它各对引物对扩增的片段,各分离物的SSCP图谱彼此均不相同。
, http://www.100md.com
3 不同BaYMV外壳蛋白基因和RNA2 70kD基因5'端705个碱基序列比较
经PCR扩增后,我们克隆了10个BaYMV分离物的外壳蛋白基因和RNA2 70kD阅读框架中5'端700个碱基片段,并进行了序列分析。结果表明,从不同分离物扩增到的上述基因或基因片段序列彼此各不相同(表3),从而证实了SSCP结果(表2)。RNA2中的这705个碱基在各个分离物之间的差异性,要比各分离物外壳蛋白基因的差异性更大(表3)。这与SSCP分析表明RNA2变异比RNA1大的结论是一致的。在经序列分析的外壳蛋白基因(包括此基因5'端上游和3'端下游共910个碱基)中,各分离物彼此存在1至20个碱基的差异,其中表3加*的差异,即杭州/南通(7)、杭州/盐城(7)、嘉善/扬州(6)和南通/盐城(1)不能用SSCP区分。同样,RNA2 70kD基因5'端的705个碱基序列中,10个分离物无一彼此相同,各分离物之间核苷酸差异为5~41个碱基,其中较小的差异,如杭州/海安(8)、扬州/盐城(5)、海安/盐城(7)均不能用SSCP加以区分(表2)
, 百拇医药
表3 中国10个BaYMV分离物之间2个基因片段核苷酸差异
Table 3 Nucleotide differences between two genome regions of 10 Chinese BaYMV isolates
分离物
Isolate
外壳蛋白基因Coat protein region(910nts)
70kD基因5'端 5'-end of 70kD region(705nts)
HA
HZ
JI
, 百拇医药
NB
NA
RU
SH
YC
YZ
HA
HZ
JI
NI
NA
RU
SH
YC
, http://www.100md.com
YZ
FU
10
16
13
13
10
15
12
11
9
14
16
15
, http://www.100md.com
16
16
34
30
13
29
HA
17
9
12
7*
11
14
, 百拇医药 7*
5
14
12
8*
18
34
31
5*
33
HZ
18
12
, http://www.100md.com
15
20
9
16
13
2
14
22
41
38
13
38
JI
15
, 百拇医药
9
12
15
10
6*
13
20
39
35
10
36
NI
12
, 百拇医药 17
10
11
11
17
34
32
7*
33
NA
9
12
1*
, 百拇医药
5
36
34
17
34
RU
17
12
18
8
32
11
SH
12
, 百拇医药
11
28
10
YC
6
29
FU:阜宁Funing,HA:海安Haian,HZ:杭州Hangzhou,JI:加善Jiashan,NI:宁波Ningbo,NA:南通Nantong,RU:如东Rudong,SH:上海Shanghai,YC:盐城Yancheng,YZ:扬州Yangzhou。
*在SSCP图谱中没有差异的组合。
*Combinations without differences in SSCP patterns.
, http://www.100md.com
4 应用酶切图谱分析区分不同BaYMV分离物
用引物N9/N10,以10个中国和3个英国BaYMV分离物RNA2的cDNA为模板,扩增705bp片段,经克隆和序列分析后,发现这10个DNA片段无一完全相同,彼此之间的差异最大的达41个碱基(表3)。根据序列差异,我们选择若干限制性内切酶包括AccⅠ、SalⅠ、AvaⅠ、CfoⅠ、DraⅠ、MspⅠ、NcoⅠ、PflmⅠ、SacⅠ和SpeⅠ,用于不同BaYMV分离物未经克隆的RT-PCR产物的直接诊断。结果表明所有中国和英国分离物均含有复合的RNA序列。这些结果也支持了复杂的SSCP图谱分析结果。有些RNA序列在各分离物之间是普遍的,有些则不然,仅在部分分离物中存在。Southern blot分析证实,所有酶切后的DNA片段来自各BaYMV分离物RNA2。
图2是各BaYMV分离物未经克隆的RT-PCR产物直接进行限制性内切酶图谱分析结果的综合。就AccⅠ和SalⅠ酶切,除嘉善、海安和南通仅含1种RNA序列外,所有其它中国和英国分离物均具有2种RNA序列,其中1种RNA序列与海安和南通分离物一样,各含有1个相同的AccⅠ和SalⅠ切点;另外1种序列和嘉善序列一样,无AccⅠ和SalⅠ切点。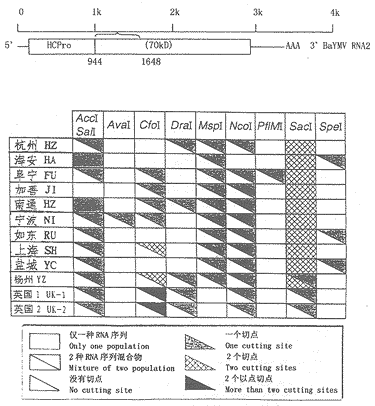
, 百拇医药
图2 限制性内切酶图谱分析中、英BaYMV分离物RNA2 70kD基因5'端区域(705个碱基)的变异
Figure 2 Variations at 5'-terminal part of 70kD region on RNA2 of Chinese and UK BaYMV isolates differentiated by restriction mapping
就AvaⅠ酶切,除宁波分离物含有2种RNA序列外,所有其他中国和英国分离物仅含有1种RNA序列,却无AvaⅠ切点。宁波的2个序列中1种含有1个AvaⅠ切点,另一种没有AvaⅠ切点。
各分离物的CfoⅠ切点更为复杂,杭州、海安、如东和盐城仅含有1种无CfoⅠ切点的序列,阜宁、嘉善、南通和宁波各自都具有2种RNA序列,其中1种无CfoⅠ切点,但另一种各自含有1个相同的CfoⅠ切点。上海、扬州也各含有2种RNA序列,1种序列无CfoⅠ切点,但另一种序列则含有2个CfoⅠ切点。UK-1和UK-2都含有混合序列,1种序列无CfoⅠ切点,另一种序列具有2个以上CfoⅠ切点。
, 百拇医药
阜宁、海安、嘉善、宁波、如东、上海和盐城分离物均各含有1种无DraⅠ切点的RNA序列,但杭州、南通、扬州、英国UK-1和UK-2都各含有2种序列,其中1种序列含1个相同的DraⅠ切点,另一种没有DraⅠ切点。
所有中国分离物一致,各自含有2种混合的序列,其中1种序列各自含有1个相同的MspⅠ切点,另一种则没有。但英国分离物均匀 无MspⅠ切点。
除了海安,所有中国和英国分离物都含有相同的NcoⅠ切点,各分离都含有2种序列,其中1种含有1个相同的NcoⅠ切点,另一种不含有NcoⅠ切点。海安分离物仅含有1种无NcoⅠ切点的序列。
除了阜宁外,所有中国和英国BaYMV分离物都相同,不含有PflmⅠ切点,但阜宁分离物具有2种序列,1种与其他分离物一样,没有PflmⅠ切点,另一种则具有1个Pfl mⅠ切点。
除了扬州分离物外,所有中国分离物具有2个相同的SacⅠ切点。不过,扬州分离物具有混合的序列,其中1种序列与其他分离物一样,具有2个SacⅠ切点,另1种序列则仅有1个SacⅠ切点。UK-1和UK-2分离物相同,都含有2种混合的序列,其中1种含有1个SacⅠ切点,另一种不含有SacⅠ切点。
, http://www.100md.com
海安、如东和盐城分离物都各含有2种序列,其中1种含有1个SpeⅠ切点,另一种序列则与其他中国分离物和英国分离物一样,都不具有SpeⅠ切点。
讨论
田间试验表明,我国BaYMV存在6个不同的株系,与已报道的2个欧洲株系和6个日本株系无一相同[7]。进而分子生物学研究表明,英国BaYMV分离物RNA1和RNA2各区域都表现出复杂的SSCP图谱,无法与田间致病性所划分的BaYMV株系在分子水平上也存在着差异,但尚不能确定导致致病性差异的分子变异[9]。本项研究表明,我国BaYMV分离物与英国BaYMV株系在分子水平存在差异,但无法简单地采用某一分子生物学技术进行中英株系的诊断和区分。
我国BaYMV分离物RNA1和RNA2都存在变异,其中RNA2的变异比RNA1更大。RNA1和RNA2在供试的10个BaYMV分离物之间序列彼此无一相同。同样,无法简单地将分子变异与不同分离物的致病性统一起来。但是,不同BaYMV分离物的分子变异也为BaYMV株系的分化和鉴定[7]提供了依据。此外,来自不同病区的BaYMV,可能存在混杂的病毒种群,甚至是混杂的病毒株系[7],复杂的SSCP图谱分析和酶切图谱分析结果正是证实了这一假设。
, 百拇医药
尽管我国不同的BaYMV分离物RNA1和RNA2无一完全相同,但根据日本和欧洲BaYMV序列设计的17对引物中,有11对能扩增全部10个供试分离物获得相应的PCR片段,只有1对引物(N29/N35)对10个供试中国分离物均不工作。另一对引物(N28/N34)只能扩增阜宁和上海分离物,不能扩增其他8个分离物。外壳蛋白基因序列分析表明,10个中国分离物的同源性高达97.8%~99.9%,RNA2上的70kD基因5'端上游705个碱基中,各分离物同源性也高达94.2%~99.7%。无疑这10个分离物不仅属于同一种病毒,而且是十分相似的。这一点也可用ELISA、Western blot、ISEM等血清学分析以及核酸杂交等方法加以证实[8]。
SSCP分析是一种区分不同病毒分离物快速、灵敏而有效的方法。不过,这项技术并不保证不同分离物序列之间微小差异的检测。本项研究表明,不同的BaYMV株系联系起来[7]。进而,病毒RNA序列差异与SSCP图谱没有简单的关系,有时,两个DNA片段之间存在2个碱基差异,就可以形成不同的SSCP图谱,相反,有时两个DNA片段之间存在8个碱基差异,也不能用SSCP进行区分。
, 百拇医药
要从分子水平诊断BaYMV不同株系,首先要测定各株系的核酸全序列,建立有活性的全长cDNA克隆,通过突变确定导致致病性变化的碱基差异,然后,根据特定的序列设计引物,才可建立快速、灵敏的株系检测诊断技术。
基金项目:欧共体ISC项目(CⅡ*-CT93-0086);国家攻关(95-015-01-026);农业部(Z96Y02);国家自然科学基金(3964002);浙江省青年科技人材专项资金(RC9604);浙江省科委(961102158)。
联系作者:陈剑平,传真:0571-6400474,E-mail:Vir@public hz.zj.cn
参考文献
1 Kashiwazaki S, Minobe Y, Omura T, et al. Nucleotide sequence of barley yellow mosaic virus RNA1:a close evolutionary relationship with potyvirus[J]. J Gen Virol, 1990, 71, 2781-1990.
, 百拇医药
2 Kashiwazaki S, Minobe Y, Hibino H, et al. Nucleotide sequence of barley yellow mosaic virus RNA2[J]. J Gen Virol, 1991, 72, 995-999.
3 Davidsin A D, Prols M, Schell J, et al. The nucleotide sequence of RNA2 of barley yellow mosaic virus[J]. J Gen Virol, 1991, 72, 989-993.
4 Peerenboom E, Prols M, Schell J, et al. The complete nucleotide sequence of RNA1 of a German isolate of barley yellow mosaic virus and its comparison with a Japanese isolate[J]. J Gen Virol, 1992, 73, 1303-1308.
, 百拇医药
5 Kashiwazaki S, Ogawa K, Usugi T, et al. Characterization of several strains of barley yellow mosaic virus[J]. Ann. Phytopath. Soc Jpn, 1989, 55: 16-25.
6 Adams M J. The distribution of barley yellow mosaic virus (BaYMV) and barley mild mosaic virus (BaMMV) in UK winter barley samples, 1987-1990[J]. Plant Pathol, 1991, 40, 53-58.
7 陈剑平,Adams M J,朱凤台,等.中国大麦黄花叶病毒(BaYMV)株系田间初步鉴定[J].中国农业科学1998,31:10-15.
, 百拇医药
8 施农农,陈剑平,应用单链构象多态性-聚合酶链反应研究我国大麦黄花叶病毒株系的分化[J].中国病毒学,1996,11(2):170-175.
9 Shi N N, Chen J P, Wilson T M A, et al. Single-strand conformation polymorphism analysis of RT-PCR products of UK isolates of barley yellow mosaic virus[J]. Virus Research, 1996, 44, 1-9.
10 Chen J P, MacFarlane S A, Wilson T M A. Detection and sequence analysis of a spontaneous deletion mutant of soil-borne wheat mosaic virus RNA2 associated with increased symptom severity[J]. Virology, 1994, 202, 921-929.
收稿日期:1998-01-20;修回日期:1998-05-05, http://www.100md.com
单位:施农农 陈剑平 浙江省农业科学院,浙江 杭州 310021;M.J.Adams 英国洛桑试验站
关键词:大麦黄花叶病毒;分子变异;单链构象多态性;序列分析;限制性切酶图谱
病毒学报990407 摘要:13个供试的中国和英国大麦黄花叶病毒(BaYMV)分离物,经RNA1和RNA2全基因组不同区域DNA片段单链构象多态性分析(SSCP),外壳蛋白基因和RNA2 70kD基因5'端705碱基序列分析,以及此705碱基DNA片段限制性内切酶图谱分析结果,它们的RNA1和RNA2彼此无一相同,其中RNA2变异比RNA1更大。由于变异十分复杂,且没有规律性,因而当前通用的分子生物学技术尚不能简单地用于BaYMV致病性分化或株系区分和检测。
中图分类号:S435.121.4 文献标识码:A 文章编号:1000-8721(1999)04-0339-09
, http://www.100md.com
MOLECULAR VARIATIONS OF CHINESE ISOLATES OF BARLEY YELLOW MOSAIC BYMOVIRUS
SHI Nong-nong1, CHEN Jian-ping1, M. J. Adams2
(1. Zhejiang Academy of Agricultural Sciences, Zhejiang,Hangzhou 310021, China;
2.IACR-Rothamsted, Harpenden, Herts, UK AL5 2JQ)
Abstract: DNA fragments amplified from the bipartite genome of thirteen Chinese and UK isolates of barley yellow mosaic bymovirus(BaYMV) by RT-PCR were analysed by single-strand conformation polymorphism(SSCP). Variations in the coat protein gene and the 5'-terminal part(c. 705bp) of the 70kD regions of RNA2 of these BaYMV isolates were also revealed by sequencing and restriction mapping. The results indicated that none of all thirteen isolates was identical each other at the molecular level. Differences in RNA2 were greater than those in RNA1. The variations were very complicated so that none of modern molecular techniques could simply correlate the variations at nucleotide level with pathogenicity or strain differentiation.
, 百拇医药
Key words: Barley yellow mosaic bymovirus; Molecular variation; Single-strand conformation polymorphism; Sequencing ;Restriction mapping
大麦黄花叶病毒(Barley yellow mosaic virus, BaYMV)是日本、中国和西北欧冬大麦的一种重要病害,是马铃薯Y病毒科大麦黄花叶病毒属(Bymovirus)的代表种,含有2个单链正链RNA基因组(RNA1为7.6kb,RNA2为3.5kb)。日本和德国BaYMV核酸全序列已测定[1-4],二者RNA1和RNA2的同源性分别为93.6%和90.4%。
BaYMV由土壤中的禾谷多粘菌Polymyxa graminis传播,轮作和农药无明显防病作用,唯一有效的防治措施是栽培抗病品种。然而,不同国家的BaYMV在致病性方面存在差异。已经表明,日本的BaYMV存在6个株系[5],欧洲的至少存在2个株系[6],我国也存在6个株系,且与日本和欧洲的株系都不同[7],已经用单链构象多态性-聚合酶链反应初步研究了不同BaYMV的变异情况[8,9]。本文应用反转录-聚合酶链反应(RT-PCR)、单链构象多态性分析(SSCP)、核酸序列分析和限制性内切酶图谱分析等分子生物学技术,进一步研究了我国BaYMV分离物的分子变异。
, http://www.100md.com
材料与方法
1 病毒分离物 1994年3月中旬和1995年3月上旬,分别从江苏省阜宁县施庄和盐城市郊区、扬州里下河地区农科所农场、如东县农科所农场、海安县农科所农场、南通市农科所农场、浙江省嘉善县良种场、浙江省农科院农场、宁波市妙山农场等地BaYMV病田,以及江苏沿海地区农科所上海病圃,采集具有典型BaYMV症状的大麦叶片,-70℃贮藏或冷冻干燥后备用。英国3个BaYMV分离物[9]由M.J.Adams提供,其中UK-1为欧洲普通株(BaYMV-1),UK-2和UK-3为抗性克服株(BaYMV-2)。
2 植物总RNA抽提 抽提方法参见文献[10]。
3 RT-PCR 分别用与BaYMV RNA1或RNA2 3'端互补的引物N11或N8(表1),以RNA1或RNA2为模板合成第一链cDNA。10μl植物总RNA,加1μl引物(500ng/μl),70℃加热10分钟,迅速在冰中冷却;然后加4μl 5×H-RT缓冲液(Life Technologies Ltd),2μl 0.1mol/L DTT,1μl 10mmol/L dNTPs和1μl水,37℃水浴2分钟;最后加1μl(200U)逆转录酶(Life Technologies Ltd),37℃水浴1小时。合成完成后,加水至总体积100μl,-20℃贮藏备用。
, 百拇医药
表1 用于BaYMV基因组扩增的引物
Table 1 Oligonucleotide primers used to amplify parts of the BaYMV genome 引物a
Primer
核苷酸位置b
Nucleotide position
序列
Sequence
温度
Tm(℃)d
, http://www.100md.com RNA1
N1(+)
30-47
5'-CAAAACGAAACAAACACA-3'
43
N26(+)
601-620
5'-CTGCAAGAGAGCTCTTCTTC-3'
55
N27(+)
1204-1223
5'-AGCGTCGTTGGGATCACTTA-3'
, http://www.100md.com
55
N28(+)
1801-1820
5'-CATGAGGGTTGGTCCAGAAT-3'
55
N29(+)
2412-2431
5'-ATTGCGTCTTGATCGACGCA-3'
55
N30(+)
3011-3030
5'-CACGCGTTGTCACTCTACAA-3'
, http://www.100md.com
55
N15(+)
3620-3638
5'-ATATGGATGCAGCCGTTGA-3'
51
N22(+)
4848-4867c
5'-TGATAGACCGGCTTGTGGGT-3'
57
N37(+)
5778-5797
, 百拇医药
5'-CTCCATCGATCCTCTCTTCT-3'
55
N12(+)
6508-6524
5'-CAAGCTGCTGATCCTCT-3'
47
N14(+)
6760-6776
5'-GATGGTAACTGGAGCGT-3'
47
N31(-)
, http://www.100md.com
800-781
5'-TTTAAGCCGCGTAGCATAGC-3'
55
N32(-)
1387-1368
5'-TCGTTGTGACTGCATCCTCA-3'
55
N33(-)
2020-2001
5'-CATCTCTGGGAGTTGCTCAA-3'
55
, 百拇医药
N34(-)
2575-2556
5'-TAAAACTCAGGAGAGCGGCA-3'
55
N35(-)
3211-3192
5'-CCGTGCATCTTCGCAAGTTT-3'
55
N36(-)
3813-3794
5'-GGTCACCTTCTTACGCCTTT-3'
, http://www.100md.com
55
N17(-)
4970-4952
5'-CCCTCAATGGCATTGTTGA-3'
51
N23(-)
6119-6100c
5'-TCGTGCCCAAACTCTTCTTC-3'
55
N16(-)
6578-6561
, 百拇医药
5'-AAGCGTGCTCCATCAGCT-3'
51
N13(-)
7407-7391
5'-GGTTTAGGTTAGTTCTG-3'
43
N11(-)
7632-7616
5'-ATTACCTTCTGGTACTC-3'
43
RNA2
, 百拇医药
N1(+)
30-47
5'-CAAAACGAAACAAACACA-3'
43
N9(+)
943-959
5'-CACAAATTGGAACTCTG-3'
43
N3(+)
1520-1536
5'-GAACCAGTTGCTAGTGG-3'
, 百拇医药
47
N4(+)
2609-2625
5'-CAAGCATCCAATCTCAG-3'
45
N5(-)
1281-1265
5'-AAACCGCCAAGATTGAT-3'
43
N10(-)
1647-1631
5'-TGGAATTTCACACGCTT-3'
, 百拇医药
43
N6(-)
1853-1837
5'-AGACAACCTCCCCAAAA-3'
45
N7(-)
2948-2932c
5'-ACCGCTAACTCTACCAT-3'
45
N8(-)
3585-3569
, 百拇医药 5'-GTCACATTTCCTGTGTA-3'
43
a.(+)病毒RNA序列,(-)病毒RNA互补序列;b.引物根据日本BaYMV序列设计(Kashiwazaki, et al.1990,1991);c.根据德国BaYMV序列设计,与日本序列有1-2个碱基差异;d.根据经验公式(A+T)×2+(C+G)×4-5计算。
a.(+)Matching, (-)Complementary to virus sequence; b. Primers designed according to sequences of Japanese BaYMV isolate (Kashiwazaki, et al. 1990, 1991); c. Designed according to the German sequence with 1 or 2 mismatches to the Japanese isolate; d. Calculated as (A+T)×2+(C+G)×4-5.
, http://www.100md.com
用表1、图1所列的引物,以cDNA1和cDNA2为模板,扩增BaYMV RNA1和RNA2 5'端至3'端各区域DNA片段。引物根据已发表的BaYMV核苷酸序列设计。50μl体积PCR反应含1μlcDNA模板,20pmol引物对,200mmol/L dNTPs,1.5mmol/L MgCl2,50mmol/L KCl,10mmol/L Tris-HCl pH8.3和2.5μl Taq DNA聚合酶(GIBCO)。经94℃变性3分钟,进行30个循环扩增,每个循环包括94℃变性1分钟,43℃~55℃退火1分钟,72℃合成1~2分钟。PCR产物经1%或1.5%琼脂糖电泳检测。

图1 BaYMV RNA1和 RNA2基因组结构及表1中所列进行RT-PCR扩增的引物
Figure 1 Genome organizations of BaYMV RNA1 and RNA2 and DNA fragments amplified by RT-PCR using the primers detailed in Table l
, 百拇医药
4 SSCP分析 参见文献[8,9]。
5 DNA克隆和序列分析 用pCRⅡ(InVitrogen公司产品)和pT7Blue(Novagen公司产品)质粒克隆PCR产物,方法见公司产品说明书。用Sequenase(Version 2.0)DNA序列分析试剂盒(USB)测定PCR克隆的核苷酸序列,方法见试剂盒说明书。
6 DNA酶切图谱分析 根据序列推测酶切位点,选择有关限制性内切酶进行酶切,在一个10μl的酶切反应中,含2μl DNA,1μl相应10×缓冲液,0.5μl酶,37℃水浴45分钟,经1%琼脂糖胶进行酶切图谱分析。有必要时,进一步用Southern blot分析[10]。
结果
1 应用RT-PCR区分不同的BaYMV分离物
, 百拇医药
以10个中国BaYMV分离物和3个英国BaYMV分离物的RNA1和RNA2的cDNA为模板,从5'端至3'端依次用不同引物对扩增不同长度的PCR片段,PCR片段大小分别为334bp至1 429bp,其中RNA1扩增区域为第31~7 632碱基,RNA2为第92~3 585碱基,均接近全长(图1)。
RNA1:用6对引物即N1/N31、N26/N32、N27/N33、N30/N36、N22/N23和N12/N13,从所有10个中国BaYMV RNA1 cDNA模板上都能扩增到预期的DNA片段,但N29/N35则不能扩增。N15/N17能从阜宁、海安、杭州、嘉善、宁波、南通、上海、盐城和扬州分离物上扩增出预期的1 352bp片段,但不能从如东分离物扩增。N14/N11除不能从嘉善分离物扩增外,从其他9个中国分离物上均能扩增到预期的873bp片段。
用引物对N30/N36扩增,所有10个中国分离物均产生一个均匀的PCR片段,但英国分离物UK-1和UK-3仅产生非常微弱的同一PCR片段,大小为803bp,与期望的片段一致,而UK-2则不产生任何产物。此PCR反应重复了4次,结果均相同。用引物对N28/N34扩增,所有3个英国分离物和阜宁、上海分离物均产生一个预期的775bp片段,而其它8个中国分离物均不扩增。用引物对N37/N16扩增后,盐城、宁波、扬州、南通、杭州、嘉善、上海分离物和所有3个英国分离物均扩增一个预期的801bp片段,而阜宁、南通和如东不产生任何产物(表2)。
, http://www.100md.com
RNA2:用6对引物即N1/N5、N19/N20、N9/N10、N3/N6、N3/N7和N4/N8,从10个供试中国BaYMV分离物RNA2模板上,均分别特异地扩增到1 252bp、922bp、705bp、334bp、1 429bp和977bp片段。经电泳分析,根据这些片段,不能区别不同的BaYMV分离物(表2)。
表2 BaYMV RNA1和RNA2 RT-PCR产物单链构象多态性分析(SSCP)图谱综合
Table 2 Summary of patterns of RT-PCR products amplified from BaYMV RNA1 and RNA2 by single-strand conformation polymorphism analysis 分离物
Isolates
RNA1
, 百拇医药
N1/N31a
N26/N32
N27/N33
N28/N34
N29/N35
N30/N36
N15/N17
N22/N23
N37/N16
N12/N13
N14/N11
771bp
, 百拇医药
687bp
817bp
775bp
800bp
803bp
1351bp
1272bp
801bp
900bp
873bp
FU
Ab
A
, 百拇医药
A
A
-c
A
A
A
-
A
A
HA
B
B
A
-
, 百拇医药
-
B
A
B
A
B
HZ
C
C
B
-
-
C
B
, 百拇医药
C
B
C
C
JI
D
A
C
-
-
B
C
A
C
, 百拇医药
D
-
NA
D
D
A
-
-
B
D
A
-
B
A
, 百拇医药
NI
E
E
A
-
-
D
A
A
D
E
B
RU
D
, 百拇医药
C
A
-
-
E
-
A
-
F
A
SH
C
A
B
, 百拇医药
B
-
F
E
A
C
G
D
YC
F
C
A
-
-
, 百拇医药
G
F
D
E
B
E
YZ
D
C
D
-
-
F
F
, 百拇医药
B
F
D
A
分离物
Isolates
RNA2
N1/N5
N19/N20
N9/N10
N3/N7
N3/N6
N4/N8
, http://www.100md.com
1252bp
922bp
705bp
1429bp
334bp
977bp
FU
A
A
A
A
A
A
, 百拇医药
HA
B
B
B
B
A
B
HZ
C
C
C
C
B
C
, http://www.100md.com
JI
D
D
D
D
C
D
NA
E
E
E
E
D
E
, http://www.100md.com
NI
F
F
B
F
E
F
RU
G
G
F
G
A
G
, http://www.100md.com
SH
H
H
G
H
F
H
YC
I
I
B
I
G
I
, http://www.100md.com
YZ
J
D
H
J
H
J
FU:阜宁Funing,HA:海安Haian,HZ:杭州Hangzhou,JI:加善Jiashan,NI:宁波Ningbo, NA:南通Nantong,RU:如东Rudong,SH:上海Shanghai,YC:盐城Yancheng,YZ:扬州Yangzhou。
a.用引物对N1/N31从RNA1扩增的DNA片段(771bp);b.同一列中相同字母表示相同SSCP图谱;c.RT-PCR扩增无产物。
, 百拇医药
a.A fragment amplified from RNA1 template with primer pair N1/N31;b.Same letter means identical SSCP pattern in one column;c.No fragment amplified by RT-PCR.
2 应用SSCP分析区分不同的BaYMV分离物
我们通过SSCP分析,鉴定了中国BaYMV分离物之间的分子差异,表2是SSCP分析结果的综合归纳。
BaYMV基因组变异比较结果表明,10个中国分离物RNA2的变异程度均比RNA1大。所有10个中国分离物N1/N5片段、N3/N7片段和N4/N8片段的SSCP图谱彼此都不相同。就SSCP图谱,N9/N10片段的,只有海安、宁波和盐城分离物相同;N3/N6片段的,只有海安、阜宁和如东分离物相同;N19/N20片段的,只有嘉善与扬州分离物相同。其它各对引物对扩增的片段,各分离物的SSCP图谱彼此均不相同。
, http://www.100md.com
3 不同BaYMV外壳蛋白基因和RNA2 70kD基因5'端705个碱基序列比较
经PCR扩增后,我们克隆了10个BaYMV分离物的外壳蛋白基因和RNA2 70kD阅读框架中5'端700个碱基片段,并进行了序列分析。结果表明,从不同分离物扩增到的上述基因或基因片段序列彼此各不相同(表3),从而证实了SSCP结果(表2)。RNA2中的这705个碱基在各个分离物之间的差异性,要比各分离物外壳蛋白基因的差异性更大(表3)。这与SSCP分析表明RNA2变异比RNA1大的结论是一致的。在经序列分析的外壳蛋白基因(包括此基因5'端上游和3'端下游共910个碱基)中,各分离物彼此存在1至20个碱基的差异,其中表3加*的差异,即杭州/南通(7)、杭州/盐城(7)、嘉善/扬州(6)和南通/盐城(1)不能用SSCP区分。同样,RNA2 70kD基因5'端的705个碱基序列中,10个分离物无一彼此相同,各分离物之间核苷酸差异为5~41个碱基,其中较小的差异,如杭州/海安(8)、扬州/盐城(5)、海安/盐城(7)均不能用SSCP加以区分(表2)
, 百拇医药
表3 中国10个BaYMV分离物之间2个基因片段核苷酸差异
Table 3 Nucleotide differences between two genome regions of 10 Chinese BaYMV isolates
分离物
Isolate
外壳蛋白基因Coat protein region(910nts)
70kD基因5'端 5'-end of 70kD region(705nts)
HA
HZ
JI
, 百拇医药
NB
NA
RU
SH
YC
YZ
HA
HZ
JI
NI
NA
RU
SH
YC
, http://www.100md.com
YZ
FU
10
16
13
13
10
15
12
11
9
14
16
15
, http://www.100md.com
16
16
34
30
13
29
HA
17
9
12
7*
11
14
, 百拇医药 7*
5
14
12
8*
18
34
31
5*
33
HZ
18
12
, http://www.100md.com
15
20
9
16
13
2
14
22
41
38
13
38
JI
15
, 百拇医药
9
12
15
10
6*
13
20
39
35
10
36
NI
12
, 百拇医药 17
10
11
11
17
34
32
7*
33
NA
9
12
1*
, 百拇医药
5
36
34
17
34
RU
17
12
18
8
32
11
SH
12
, 百拇医药
11
28
10
YC
6
29
FU:阜宁Funing,HA:海安Haian,HZ:杭州Hangzhou,JI:加善Jiashan,NI:宁波Ningbo,NA:南通Nantong,RU:如东Rudong,SH:上海Shanghai,YC:盐城Yancheng,YZ:扬州Yangzhou。
*在SSCP图谱中没有差异的组合。
*Combinations without differences in SSCP patterns.
, http://www.100md.com
4 应用酶切图谱分析区分不同BaYMV分离物
用引物N9/N10,以10个中国和3个英国BaYMV分离物RNA2的cDNA为模板,扩增705bp片段,经克隆和序列分析后,发现这10个DNA片段无一完全相同,彼此之间的差异最大的达41个碱基(表3)。根据序列差异,我们选择若干限制性内切酶包括AccⅠ、SalⅠ、AvaⅠ、CfoⅠ、DraⅠ、MspⅠ、NcoⅠ、PflmⅠ、SacⅠ和SpeⅠ,用于不同BaYMV分离物未经克隆的RT-PCR产物的直接诊断。结果表明所有中国和英国分离物均含有复合的RNA序列。这些结果也支持了复杂的SSCP图谱分析结果。有些RNA序列在各分离物之间是普遍的,有些则不然,仅在部分分离物中存在。Southern blot分析证实,所有酶切后的DNA片段来自各BaYMV分离物RNA2。
图2是各BaYMV分离物未经克隆的RT-PCR产物直接进行限制性内切酶图谱分析结果的综合。就AccⅠ和SalⅠ酶切,除嘉善、海安和南通仅含1种RNA序列外,所有其它中国和英国分离物均具有2种RNA序列,其中1种RNA序列与海安和南通分离物一样,各含有1个相同的AccⅠ和SalⅠ切点;另外1种序列和嘉善序列一样,无AccⅠ和SalⅠ切点。

, 百拇医药
图2 限制性内切酶图谱分析中、英BaYMV分离物RNA2 70kD基因5'端区域(705个碱基)的变异
Figure 2 Variations at 5'-terminal part of 70kD region on RNA2 of Chinese and UK BaYMV isolates differentiated by restriction mapping
就AvaⅠ酶切,除宁波分离物含有2种RNA序列外,所有其他中国和英国分离物仅含有1种RNA序列,却无AvaⅠ切点。宁波的2个序列中1种含有1个AvaⅠ切点,另一种没有AvaⅠ切点。
各分离物的CfoⅠ切点更为复杂,杭州、海安、如东和盐城仅含有1种无CfoⅠ切点的序列,阜宁、嘉善、南通和宁波各自都具有2种RNA序列,其中1种无CfoⅠ切点,但另一种各自含有1个相同的CfoⅠ切点。上海、扬州也各含有2种RNA序列,1种序列无CfoⅠ切点,但另一种序列则含有2个CfoⅠ切点。UK-1和UK-2都含有混合序列,1种序列无CfoⅠ切点,另一种序列具有2个以上CfoⅠ切点。
, 百拇医药
阜宁、海安、嘉善、宁波、如东、上海和盐城分离物均各含有1种无DraⅠ切点的RNA序列,但杭州、南通、扬州、英国UK-1和UK-2都各含有2种序列,其中1种序列含1个相同的DraⅠ切点,另一种没有DraⅠ切点。
所有中国分离物一致,各自含有2种混合的序列,其中1种序列各自含有1个相同的MspⅠ切点,另一种则没有。但英国分离物均匀 无MspⅠ切点。
除了海安,所有中国和英国分离物都含有相同的NcoⅠ切点,各分离都含有2种序列,其中1种含有1个相同的NcoⅠ切点,另一种不含有NcoⅠ切点。海安分离物仅含有1种无NcoⅠ切点的序列。
除了阜宁外,所有中国和英国BaYMV分离物都相同,不含有PflmⅠ切点,但阜宁分离物具有2种序列,1种与其他分离物一样,没有PflmⅠ切点,另一种则具有1个Pfl mⅠ切点。
除了扬州分离物外,所有中国分离物具有2个相同的SacⅠ切点。不过,扬州分离物具有混合的序列,其中1种序列与其他分离物一样,具有2个SacⅠ切点,另1种序列则仅有1个SacⅠ切点。UK-1和UK-2分离物相同,都含有2种混合的序列,其中1种含有1个SacⅠ切点,另一种不含有SacⅠ切点。
, http://www.100md.com
海安、如东和盐城分离物都各含有2种序列,其中1种含有1个SpeⅠ切点,另一种序列则与其他中国分离物和英国分离物一样,都不具有SpeⅠ切点。
讨论
田间试验表明,我国BaYMV存在6个不同的株系,与已报道的2个欧洲株系和6个日本株系无一相同[7]。进而分子生物学研究表明,英国BaYMV分离物RNA1和RNA2各区域都表现出复杂的SSCP图谱,无法与田间致病性所划分的BaYMV株系在分子水平上也存在着差异,但尚不能确定导致致病性差异的分子变异[9]。本项研究表明,我国BaYMV分离物与英国BaYMV株系在分子水平存在差异,但无法简单地采用某一分子生物学技术进行中英株系的诊断和区分。
我国BaYMV分离物RNA1和RNA2都存在变异,其中RNA2的变异比RNA1更大。RNA1和RNA2在供试的10个BaYMV分离物之间序列彼此无一相同。同样,无法简单地将分子变异与不同分离物的致病性统一起来。但是,不同BaYMV分离物的分子变异也为BaYMV株系的分化和鉴定[7]提供了依据。此外,来自不同病区的BaYMV,可能存在混杂的病毒种群,甚至是混杂的病毒株系[7],复杂的SSCP图谱分析和酶切图谱分析结果正是证实了这一假设。
, 百拇医药
尽管我国不同的BaYMV分离物RNA1和RNA2无一完全相同,但根据日本和欧洲BaYMV序列设计的17对引物中,有11对能扩增全部10个供试分离物获得相应的PCR片段,只有1对引物(N29/N35)对10个供试中国分离物均不工作。另一对引物(N28/N34)只能扩增阜宁和上海分离物,不能扩增其他8个分离物。外壳蛋白基因序列分析表明,10个中国分离物的同源性高达97.8%~99.9%,RNA2上的70kD基因5'端上游705个碱基中,各分离物同源性也高达94.2%~99.7%。无疑这10个分离物不仅属于同一种病毒,而且是十分相似的。这一点也可用ELISA、Western blot、ISEM等血清学分析以及核酸杂交等方法加以证实[8]。
SSCP分析是一种区分不同病毒分离物快速、灵敏而有效的方法。不过,这项技术并不保证不同分离物序列之间微小差异的检测。本项研究表明,不同的BaYMV株系联系起来[7]。进而,病毒RNA序列差异与SSCP图谱没有简单的关系,有时,两个DNA片段之间存在2个碱基差异,就可以形成不同的SSCP图谱,相反,有时两个DNA片段之间存在8个碱基差异,也不能用SSCP进行区分。
, 百拇医药
要从分子水平诊断BaYMV不同株系,首先要测定各株系的核酸全序列,建立有活性的全长cDNA克隆,通过突变确定导致致病性变化的碱基差异,然后,根据特定的序列设计引物,才可建立快速、灵敏的株系检测诊断技术。
基金项目:欧共体ISC项目(CⅡ*-CT93-0086);国家攻关(95-015-01-026);农业部(Z96Y02);国家自然科学基金(3964002);浙江省青年科技人材专项资金(RC9604);浙江省科委(961102158)。
联系作者:陈剑平,传真:0571-6400474,E-mail:Vir@public hz.zj.cn
参考文献
1 Kashiwazaki S, Minobe Y, Omura T, et al. Nucleotide sequence of barley yellow mosaic virus RNA1:a close evolutionary relationship with potyvirus[J]. J Gen Virol, 1990, 71, 2781-1990.
, 百拇医药
2 Kashiwazaki S, Minobe Y, Hibino H, et al. Nucleotide sequence of barley yellow mosaic virus RNA2[J]. J Gen Virol, 1991, 72, 995-999.
3 Davidsin A D, Prols M, Schell J, et al. The nucleotide sequence of RNA2 of barley yellow mosaic virus[J]. J Gen Virol, 1991, 72, 989-993.
4 Peerenboom E, Prols M, Schell J, et al. The complete nucleotide sequence of RNA1 of a German isolate of barley yellow mosaic virus and its comparison with a Japanese isolate[J]. J Gen Virol, 1992, 73, 1303-1308.
, 百拇医药
5 Kashiwazaki S, Ogawa K, Usugi T, et al. Characterization of several strains of barley yellow mosaic virus[J]. Ann. Phytopath. Soc Jpn, 1989, 55: 16-25.
6 Adams M J. The distribution of barley yellow mosaic virus (BaYMV) and barley mild mosaic virus (BaMMV) in UK winter barley samples, 1987-1990[J]. Plant Pathol, 1991, 40, 53-58.
7 陈剑平,Adams M J,朱凤台,等.中国大麦黄花叶病毒(BaYMV)株系田间初步鉴定[J].中国农业科学1998,31:10-15.
, 百拇医药
8 施农农,陈剑平,应用单链构象多态性-聚合酶链反应研究我国大麦黄花叶病毒株系的分化[J].中国病毒学,1996,11(2):170-175.
9 Shi N N, Chen J P, Wilson T M A, et al. Single-strand conformation polymorphism analysis of RT-PCR products of UK isolates of barley yellow mosaic virus[J]. Virus Research, 1996, 44, 1-9.
10 Chen J P, MacFarlane S A, Wilson T M A. Detection and sequence analysis of a spontaneous deletion mutant of soil-borne wheat mosaic virus RNA2 associated with increased symptom severity[J]. Virology, 1994, 202, 921-929.
收稿日期:1998-01-20;修回日期:1998-05-05, http://www.100md.com