HCV 5′NCR 和结构蛋白编码序列的拼接及在pLNSX中的克隆
作者:曹开源 冯炼强 刘亚光 付涌水 吕凌
单位:(中山医科大学 免疫学教研室, 广东 广州 510089)
关键词:克隆,分子;聚合酶链反应;肝炎病毒,丙型
中国病理生理杂志000910
[摘 要] 目的:构建丙型肝炎病毒(HCV) 5′末端非编码区(5′NCR)和结构蛋白编码基因序列逆转录病毒重组体,用于探索控制HCV感染的新途径和基因治疗。方法:对多株HCV核酸序列进行同源性比较设计引物,逆转录聚合酶链式反应(RT-PCR)扩增5′NCR、C、E1和E2/NS1 4个编码区共5个片段,分别克隆。以连接聚合酶链反应(PCR)将这5个片段拼接为一连续的长2547 nt的片段,含HCV完整的5′NCR和全部的结构蛋白编码序列。将此序列插入pGEM-Zf(+)载体,与逆转病毒pLNSX载体中,转化大肠杆菌DH5a、转化菌落经酶切、PCR和Southern杂交鉴定。结果:通过RT-PCR和连接聚合酶链式反应(PCR)扩增出2547 nt含HCV完整的5′NCR和全部的结构蛋白编码序列,将此序列与pGEM-Zf(+)重组得重组体pHC2547,与逆转录病毒载体pLNSX重组得pLHC。结论:成功构建了HCV逆转病毒重组体,以利于HCV的胞内基因表达调控研究,更为探索HCV分子致病机制和转基因动物及基因治疗提供可靠的物质基础。
, http://www.100md.com
[中图分类号] R512.6+3 [文献标识码] A
[文章编号] 1000-4718(2000)09-0809-05
Ligation of the HCV 5'NCR, C, E1, E2/NS1 sequences
and its cloning into pLNSX
CAO Kai-yuan, FENG Lian-qiang
(Department of Immunology ,Guangzhou 510089, China)
LIU Ya-guang
(Department of Nephrology of the first Affiliated Hospital, ,Guangzhou 510089, China)
, 百拇医药
FU Yong-shui, LU Ling
(The Research Center of Molecular Medicine, Sun Yat Sen University of Medical Sciences ,Guangzhou 510089, China)
[Abstract] AIM:To construct retrovirus recombinant HCV/ pLNSX(pLHC), which is used to investigate new ways to control HCV infection and gene therapy. METHODS and RESULTS: Primers with their sequences homologous to multiple strains of HCV had been designed, synthesized and used in RT-PCR to amplify 5 fragments from 4 regions,5'NCR,C,E1 and E2/NS1.The products had been cloned, restrictively cut aside, and overlappingly amplified to ligate a consecutive sequence of 2547 bp in length that contains the whole HCV 5'NCR and all the structural protein coding regions. The sequence was then inserted into the vector pGEM-3Zf (+) yielding a recombinant pHC2547, and into the retrovirus pLNSX yielding a pLHC .Both plasmid DNA were analyzed by PCR, Southern blot, and enzymatic cut. CONCLUSION: Retrovirus recombinant HCV/pLNSX (pLHC) constructed successfully is useful to study both the regulation of intracellular HCV gene expression and the pathogenesis of HCV infection, as well as the molecular foundation of related transgene animals and gene therapy.
, 百拇医药
[MeSH] Cloning; Molecular; Polymerase chain reaction; Hepatitis C virus
丙型肝炎病毒(hepatitis C virus, HCV)基因组长9600 nt,其中5’末端非编码区(5′ non-coding region,5’NCR)长324 核苷酸(nucleotides, nt),核壳蛋白编码区(C)和包膜蛋白1编码区(E1)分别为573和576 nt,包膜 蛋白2编码区(E2/NS1)长1140 nt,这4 段序列共长约2600 nt[1]。本文应用聚合酶链式反应(polymerase chain reaction, PCR)和分子克隆技术将之拼接成为连续序列,再分别正向插入pGEM-3Zf(+)和pLNSX载体中,构建了pHC2547和pLHC重组体,以利于研究HCV的5′NCR序列对后续结构蛋白基因表达的调控作用, 更为探索HCV分子致病机制和转基因动物及基因治疗提供可靠的物质基础。
, 百拇医药
材 料 和 方 法
一、 HCV基因引物
比较多株HCV序列自行进行设计,以ABI391型全自动脱氧核糖核酸(deoxyribonucleic acid, DNA)合成仪人工合成(表1)
二、 核糖核酸(ribonucleic acid. RNA)提取、逆转录、PCR扩增, 同前文[3]。
三、逆转录聚合酶链式反应(reverse transcription-polymerase chain reaction, RT-PCR) 产物克隆
以pUC18为载体,对引物NC1和NC2的扩增产物作钝端克隆,其余4对引物的扩增产物作粘端定向克隆。
四、 基因拼接与亚克隆
, http://www.100md.com
以合适的内切酶切出克隆基因后用 expandTM high fedelity PCR system (Boehringer-Mannheim公司产品)以连接PCR方法两两连接,连接物以pUC18克隆。然后重复以上步骤,迄至5个片段依均序完成拼接,最后的拼接产物以pGEM-3Zf(+)克隆。
五、 拼接基因以pLNSX 克隆
Hind Ⅲ酶切 pLNSX载体,Pst I与EcoRI切出pGEM-3Zf(+)中克隆的HCV基因,然后两者作钝端克隆。
六、 重组体筛选
挑取单个菌落过夜培养,常规提取质粒DNA 后酶切和电泳鉴定或以地高辛素标记的HCV目的基因作斑点杂交,杂交阳性克隆再作限制性酶切和PCR鉴定。
表1 HCV基因的引物序列
, http://www.100md.com
Tab 1 Primer sequences of HCV gene Prime
Nucleotide location
Polarity
Sequence(5’——3’)
NC1
-324— -304
+
GGCGACACTCCACCATAGATC
NC2
-1—-21
-
, 百拇医药
GGTGCACGGTCTACGAGACCT
C1
-71—-44
+
GCGAATTCCCTTGTGGTACTGCCTGATA
C2
479—507
-
CAGGATCCCTGTTGCATAGTTCACGCCG
E1
469—493
, http://www.100md.com +
TTCTGCAGGACGGCGTGAACTATGC
E2
964—986
-
GTAGGATCCCAGTTCATCATCAT
E3
959—981
+
TGGAATTCGATGATGAACTGGTC
E4
1741—1763
, 百拇医药
-
TTTCTGCAGCAATCCGTGGGGCA
E5
1444—1467
+
CAGAAGCTTTATTGCTGGCACTAC
E6
2200—2223
-
TATCTGCAGCATCATCCACAAGCA
Nucleotide location:Site A of the first ATG on Chinese Hebei strain[2]recognized location 1.GAATTC:EcoRI recognition sequence. GGATCC:BamH I recognition sequence. CTGCAG:Pst I recognition sequence. AAGCTT:HindⅢ recognition sequence.结 果
, http://www.100md.com
一、RT-PCR 产物克隆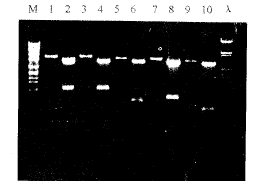
Fig 1 Fragmental clonig of PCR products.
M: SppI/EcoRI DNA maker
1. linear pHC780;2. pHC780/HindIII+PstI;
3. linear pHC805;4. pHC805/EcoRI +PstI;
5. linear pHC 528;6. pHC528/BamH I+PstI;
7. linear pHC578;8. pHC578/EcoRI+BamHI;
, 百拇医药
9. linear pHC324;10. pHC324/EcoRI+BamHI;
λ. λDNA/EcoRI +BamHI.
图1 PCR产物分段克隆
NC1 和NC2 引物扩增的产物长324 bp,钝端正向插入pUC18的Sma I 切点重组为pHC324。C1和C2 引物扩增的产物长578 bp,粘端反向插入pUC18的EcoR I和BamH I双切点重组为pHC578。E1和E2引物扩增的产物长528 bp,粘端正向插入pUC18的BamH I和Pst I双切点重组为pHC528。E3和E4引物扩增的产物长805 bp,粘端反向插入pUC18的EcoRI 和Pst I双切点重组为pHC805。E5和E6引物扩增的产物长780 bp,粘端正向插入pUC18的HindⅢ和PstI双切点重组为pHC780(图1)。
, 百拇医药 二、第一步拼接克隆
分别以EcoRI与BamHI双切pHC578和以Pst I与BamH I双切pHC528,切出的克隆基因以连接PCR拼接后粘端反向插入pUC18的EcoRI 和BamHI双切点重组为pHC1050。分别以EcoRI与PstI双切pHC805 和以HindⅢ与Pst I 双切pHC780,切出的克隆基因以连接PCR拼接后粘端反向插入pUC18的EcoRI 与Pst I 双切点重组为pHC1260(图2)。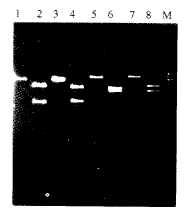
Fig 2 Splicing cloning of PCR products.
1. linear pHC1050;2. pHC1050/EcoRI+BamHI;
3. linear pHC1260; 4. pHC1260/EcoRI+PstI;
, 百拇医药
5. linear pHC2294; 6. pHC2294/EcoRI+PstI;
7. linear pHC2547; 8. pHC2547/EcoRI +PstI;
M.SppI/EcoRI DNA Maker.
图2 PCR产物拼接克隆
三、第二步拼接克隆
分别以EcoRI与BamHI 双切pHC1050和以EcoRI与Pst I双切pHC 1260,切出的克隆基因以连接PCR拼接后粘端反向插入pUC18 的EcoRI与Pst I双切点重组为pHC2294(图2),pHC2294含HCV几乎全部的结构蛋白编码序列。
四、第三步拼接克隆
, 百拇医药
以EcoRI与Pst I 双切pHC2294和以EcoRI与BamHI双切pHC324,切出的克隆基因以连接PCR拼接后钝端正向插入pGEM-3Zf(+)的Sma I切点重组为pHC2547(图2),pHC2547含HCV全部5'NCR序列和几乎全部的结构蛋白编码序列。
五、pHC2547鉴定
图3可见,由324 bp、578 bp、528 bp、805 bp 和780 bp这5个片段拼接而来的目的基因电泳位置(第8 电泳道)略低于对照分子量标准(M泳道)的第6条带(2.7 kb ),与目的基因的理论长度2547 bp相符;pGEM-3Zf(+)为3199 bp,迁移位置(第7泳道)低于对照标准的第5条带(3.5 kb);当与目的基因重组后分子量增大,迁移位置上升至接近于对照标准的第3条带(6.0 kb),与pHC2547的理论长度5746 bp相符。在PGEM-3Zf(+)多克隆位点各有1个EcoR I、Xba I、BamH I和Sac I切点,在目的基因第1345 nt和2238 nt位置依次有1个BamH I和Sac I切点,故pHC2547可被EcoR I 和Xba I 切为2个片段,其大小1条相当于pGEM-3Zf(+)空载体,1条相当于完整的克隆目的基因(第5泳道 );BamH I 也切出两条带(第4泳道),1条位于1.4 kb 和1.15 kb标准带之间,另1条略低于4.8 kb,与理论计算值4541 bp和1205 bp相符;Sac I切出的两条带(第3 泳道),1条小于2.7 kb但大于1.9 kb,另1条相当于3.5 kb,与理论计算值2238 bp和3508 bp也相符。对pHC2547以NC1和E6引物作长片段PCR扩增,得产物分子量与目的基因相若(第2泳道);以NC1和E2引物扩增,产物迁移位置稍低于1.4 kb(第1泳道),与实际大小1300 bp相符。以C1/E4引物经PCR制备地高辛素标记的1834 bp的HCV-cDNA探针对图3电泳模式作Southern 转移杂交结果见(图4)。第3和第5泳道的2条带中都是分子量小者阳性而大者阴性,说明小者含有而大者不含有探针同源序列;其余泳道则与电泳模式一致。因此拼接基因得以鉴定。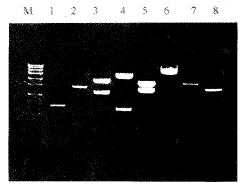
, 百拇医药
Fig 3 Identification of pHC2547 clone.
M:SppI/EcoRI NDA maker;
1.product of pHC2547 DNA amplified by prime NC1 and E2(1300 bp);
2.product of pHC2547 NDA amplified by prime NC1 and E6(2547bp);
3.pHC2547/SacI; 4.pHC2547/BamHI;
5.pHC2547/EcoRI+XbaI; 6.linear pHC2547;
7.linear pGEM-3Zf(+);
, 百拇医药
8.splicing target gene(2547bp).
图3 pHC2547 克隆鉴定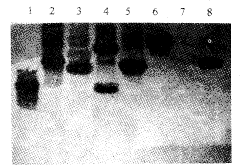
Fig 4 Mapping of Fig3 by Southern hybridization.
图4 图3的Southern杂交结果
六、逆转录病毒重组体pLHC的构建及鉴定
HindⅢ酶切pLNSX为载体,PstI与EcoRI切出pHC2547中的克隆序列为目的基因,经Klenow酶补平未端后以T4 DNA连接酶连接,连接物转化DH5a钙化菌并接种Luria-Bertani培养基平板。挑取单个菌落过夜培养,提取质粒DNA作斑点杂交,筛选阳性克隆菌。共筛选162个菌落,其中29个阳性,DNA酶切,发现有9个克隆其插入片段大小正确,4个为正向,5个为反向。其中1个正向者命名为pLHC,其酶切鉴定结果示(图5)。与对照DNA marker(M泳道)相比,目的基因(8泳道)略小于2.7 kb,载体(7泳道)约为6.0 kb,两者重组后(6泳道)大于8.0 kb,与实际大小2561 bp、6149 bp和8715 bp相符。以DNASIS软件模拟pLHC重组可见:Pst I与EcoR I切出目的基因补平末端后长2561 bp,上游残留pGEM 3Zf(+)的18 bp,下游4 nt则被Pst I切除;与pLNSX重组后长8715 bp,其中3167-5709 nt为HCV序列,3149-3166 nt为pGEM 3Zf(+)残留。模拟酶切提示:6050 nt有XbaI切点,3859 nt和5718 nt有Cla I切点,2798 nt和4507 nt有BamH I切点,1132 nt、1140 nt、1316 nt、1838 nt、2761 nt和7969 nt有Pst I切点,2 nt、1615 nt有EcoR I切点。酶切pLHC结果Xba I使之线性化(6泳道),Cla I切出6823 bp和1860 bp 2个片段(5泳道),BamH I切出6974 bp和1709 bp两个片段(4泳道),Pst I切出5208 bp和1846 bp、923 bp、522 bp和276 bp 5个片段(3泳道),EcoR I切出7070 bp和1613 bp 2个片段(2泳道)。而资作对照的EcoR I切空载体则切出4536 bp和1613 bp 2个片段(1泳道).实际结果与计算机推导完全一致。 (图6) 为pLHC的PCR鉴定结果,引物见前文[2],都扩增出预期大小的产物。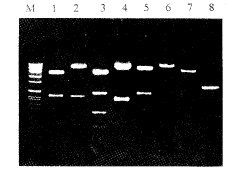
, http://www.100md.com
Fig 5 Identifieation of pLHC by enzymes.
M. SppI/EcoR I DNA marker;
1. pLNSX/EcoR I; 2. pLHC/EcoR I;
3. pLHC/Pst I;4. pLHC/BamH I;
5. pLHC/Cla I; 6. pLHC/Xba I;
7. pLNSX/Hind III; 8. pHC2547/EcoR I +Pst I.
图5 pLHC酶切鉴定
, 百拇医药
Fig 6 Identification of pLHC DNA by PCR.
M. SppI/EcoRI DNA maker;
1.product amplified by Prime NC1 and E6(2547bp);
2.product amplified by Prime E3 and E6(1265bp);
3.product amplified by Prime NC3 and E2(1260bp);
4.product amplified by prime C1 and E6(2294bp);
5.product amplified by prime NC1 and E2(1310bp);
, 百拇医药
6.product amplified by prime E1 and E2 (528bp);
7.product amplified by prime C1 and C2(578bp);
8.product amplified by prime NC1 and NC2(324bp).
图6 pLHC DNA的PCR鉴定
讨 论
HCV是第一个以反向生物学方法阐明的人类病毒。其病毒颗粒虽未确认,但基因序列却已克隆。HCV全基因组长9600 nt,携一个开放读框(open reading frame,ORF),后者又划分为若干基因区。由5′→3′方向排序依次为C、E1、E2/NS1、NS2-NS5共7个区。其中C区编码病毒的核壳蛋白,E1区编码病毒的包膜蛋白,E2/NS1区则可能编码HCV的第二种包膜蛋白,这3个区包含HCV的全部结构蛋白编码基因[1]。ORF上游还有一个保守的非编码区,称为5′NCR,这是调控HCV基因组复制和表达的关键序列[4]。
, http://www.100md.com
HCV 基因表达已有成熟的结果。重组的HCV 核壳蛋白一直以来都是ELISA 检测血清抗HCV 免疫诊断试剂的关键抗原,其来源包括原核表达、酵母表达[5]、昆虫细胞表达[6]和哺乳动物细胞表达[7]。重组E蛋白曾是研究HCV基因重组疫苗的热点,尽管最终归于失败,但却以此帮助了解到,HCV 具有准种系病毒(QUASISPECIES)这一新的特点[8]。有关非结构蛋白基因的表达也有不少报道。HCV的5′NCR序列对HCV基因复制和表达有极其重要的调控作用,它不仅含有病毒基因复制所需的核糖体进入位点,而且还有其它一些重要的顺式调控元件[4]。体外表达实验中,反义寡核酸选择性的封阻其某一区域可强力抑制后续序列的基因表达,但还缺乏细胞内和体内的实验依据[9]。
逆转录病毒载体能有效介导外源基因整合至宿主细胞染色体中,这一效率高达100%,介入的基因也几乎均能获得有效表达。此优点使逆转录病毒载体成为介导人体基因治疗的重要手段之一[10]。本文为研究HCV 的NCR序列对其结构蛋白编码基因表达的调控作用,拼接重组了包括完整5′NCR和全部结构蛋白编码基因的连续序列作为目的基因,顺向插入逆转录病毒载体pLNSX的SV40启动子下游,构建了pLHC重组体,这为进一步研究细胞内基因表达调控和转基因动物及基因治疗提供了可靠的物质基础。
, http://www.100md.com
[基金项目] 国家自然科学基金资助(No.39570285 )
Tel: 020-87333619; E-mail:kycao@gzsums. edu.cn
[参 考 文 献]
[1] 金冬雁. 丙型肝炎病毒分子生物学研究动态和发展趣向[M]. 见:侯云德,金冬雁,主编. 现代分子病毒学选论. 北京:科学出版社, 1994. 244-260.
[2] 毕胜利,白宪鹤,刘崇柏,等.我国丙型肝炎病毒河北株基因组的克隆及cDNA全序列的测序与分析[J]. 中华实验和临床病毒学杂志,1992,6(4):425-434.
[3] 吕 凌,王 斌. HCV 5′NCR 序列克隆和转录重组质粒构建[J]. 中山医科大学学报,1996,17(3):166-170
, 百拇医药
[4] Fukushi S, Katayama K, Kurihara C, et al. Complete 5’ noncoding region is necessary for the efficient internal initiation of hepatitis C virus RNA[J] . Biochem Biophys Res Commun,1994,199(2):425-432.
[5] Fillice G, Patruno S, Campisi D , et al. Specificity and sensitivity of 3rd generation EIA for detection of HCV antibodies among introvenous drug-users[J]. Microbiologica,1993,16(1):35-42.
[6] Hirowatari Y, Hijikata M, Tanji Y , et al . Expression and processing of putative nonstructural proteins of hepatitis C virus in insect cells using baculovirus vector[J]. Virus Res ,1995,35(1):43-61.
, 百拇医药
[7] Chang SC, Yen JH , Kang HY,et al . Nuclear localization signals in the core protein of hepatitis C virus[J]. Biochem Biophys Res Commun , 1994,205(2):1284-1290.
[8] Bukh J, Miller RH, Purcell RH. Genetic heterogeneity of hepatitis C virus: quasispecies and genotypes[J]. Semin liver Dis, 1995,15(1):41-63.
[9] Wakiba T, Wands JR. Specific inhibition of hepatitis C virus expression by antisense oligonucleotides[J]. J Bio Chem, 1994, 269(9): 14205-14210.
[10] Aoki K,Yochida T, Sugimura T, et al. Liposome-mediated in vivo gene transfer of antisense K-ras construct inhibits pancreatic tumor dissemination in the murine peritoneal cavity[J]. Cancer Res, 1995,55(17): 3810-3816.
[收稿日期] 2000-04-17 [修回日期] 2000-07-20, 百拇医药
单位:(中山医科大学 免疫学教研室, 广东 广州 510089)
关键词:克隆,分子;聚合酶链反应;肝炎病毒,丙型
中国病理生理杂志000910
[摘 要] 目的:构建丙型肝炎病毒(HCV) 5′末端非编码区(5′NCR)和结构蛋白编码基因序列逆转录病毒重组体,用于探索控制HCV感染的新途径和基因治疗。方法:对多株HCV核酸序列进行同源性比较设计引物,逆转录聚合酶链式反应(RT-PCR)扩增5′NCR、C、E1和E2/NS1 4个编码区共5个片段,分别克隆。以连接聚合酶链反应(PCR)将这5个片段拼接为一连续的长2547 nt的片段,含HCV完整的5′NCR和全部的结构蛋白编码序列。将此序列插入pGEM-Zf(+)载体,与逆转病毒pLNSX载体中,转化大肠杆菌DH5a、转化菌落经酶切、PCR和Southern杂交鉴定。结果:通过RT-PCR和连接聚合酶链式反应(PCR)扩增出2547 nt含HCV完整的5′NCR和全部的结构蛋白编码序列,将此序列与pGEM-Zf(+)重组得重组体pHC2547,与逆转录病毒载体pLNSX重组得pLHC。结论:成功构建了HCV逆转病毒重组体,以利于HCV的胞内基因表达调控研究,更为探索HCV分子致病机制和转基因动物及基因治疗提供可靠的物质基础。
, http://www.100md.com
[中图分类号] R512.6+3 [文献标识码] A
[文章编号] 1000-4718(2000)09-0809-05
Ligation of the HCV 5'NCR, C, E1, E2/NS1 sequences
and its cloning into pLNSX
CAO Kai-yuan, FENG Lian-qiang
(Department of Immunology ,Guangzhou 510089, China)
LIU Ya-guang
(Department of Nephrology of the first Affiliated Hospital, ,Guangzhou 510089, China)
, 百拇医药
FU Yong-shui, LU Ling
(The Research Center of Molecular Medicine, Sun Yat Sen University of Medical Sciences ,Guangzhou 510089, China)
[Abstract] AIM:To construct retrovirus recombinant HCV/ pLNSX(pLHC), which is used to investigate new ways to control HCV infection and gene therapy. METHODS and RESULTS: Primers with their sequences homologous to multiple strains of HCV had been designed, synthesized and used in RT-PCR to amplify 5 fragments from 4 regions,5'NCR,C,E1 and E2/NS1.The products had been cloned, restrictively cut aside, and overlappingly amplified to ligate a consecutive sequence of 2547 bp in length that contains the whole HCV 5'NCR and all the structural protein coding regions. The sequence was then inserted into the vector pGEM-3Zf (+) yielding a recombinant pHC2547, and into the retrovirus pLNSX yielding a pLHC .Both plasmid DNA were analyzed by PCR, Southern blot, and enzymatic cut. CONCLUSION: Retrovirus recombinant HCV/pLNSX (pLHC) constructed successfully is useful to study both the regulation of intracellular HCV gene expression and the pathogenesis of HCV infection, as well as the molecular foundation of related transgene animals and gene therapy.
, 百拇医药
[MeSH] Cloning; Molecular; Polymerase chain reaction; Hepatitis C virus
丙型肝炎病毒(hepatitis C virus, HCV)基因组长9600 nt,其中5’末端非编码区(5′ non-coding region,5’NCR)长324 核苷酸(nucleotides, nt),核壳蛋白编码区(C)和包膜蛋白1编码区(E1)分别为573和576 nt,包膜 蛋白2编码区(E2/NS1)长1140 nt,这4 段序列共长约2600 nt[1]。本文应用聚合酶链式反应(polymerase chain reaction, PCR)和分子克隆技术将之拼接成为连续序列,再分别正向插入pGEM-3Zf(+)和pLNSX载体中,构建了pHC2547和pLHC重组体,以利于研究HCV的5′NCR序列对后续结构蛋白基因表达的调控作用, 更为探索HCV分子致病机制和转基因动物及基因治疗提供可靠的物质基础。
, 百拇医药
材 料 和 方 法
一、 HCV基因引物
比较多株HCV序列自行进行设计,以ABI391型全自动脱氧核糖核酸(deoxyribonucleic acid, DNA)合成仪人工合成(表1)
二、 核糖核酸(ribonucleic acid. RNA)提取、逆转录、PCR扩增, 同前文[3]。
三、逆转录聚合酶链式反应(reverse transcription-polymerase chain reaction, RT-PCR) 产物克隆
以pUC18为载体,对引物NC1和NC2的扩增产物作钝端克隆,其余4对引物的扩增产物作粘端定向克隆。
四、 基因拼接与亚克隆
, http://www.100md.com
以合适的内切酶切出克隆基因后用 expandTM high fedelity PCR system (Boehringer-Mannheim公司产品)以连接PCR方法两两连接,连接物以pUC18克隆。然后重复以上步骤,迄至5个片段依均序完成拼接,最后的拼接产物以pGEM-3Zf(+)克隆。
五、 拼接基因以pLNSX 克隆
Hind Ⅲ酶切 pLNSX载体,Pst I与EcoRI切出pGEM-3Zf(+)中克隆的HCV基因,然后两者作钝端克隆。
六、 重组体筛选
挑取单个菌落过夜培养,常规提取质粒DNA 后酶切和电泳鉴定或以地高辛素标记的HCV目的基因作斑点杂交,杂交阳性克隆再作限制性酶切和PCR鉴定。
表1 HCV基因的引物序列
, http://www.100md.com
Tab 1 Primer sequences of HCV gene Prime
Nucleotide location
Polarity
Sequence(5’——3’)
NC1
-324— -304
+
GGCGACACTCCACCATAGATC
NC2
-1—-21
-
, 百拇医药
GGTGCACGGTCTACGAGACCT
C1
-71—-44
+
GCGAATTCCCTTGTGGTACTGCCTGATA
C2
479—507
-
CAGGATCCCTGTTGCATAGTTCACGCCG
E1
469—493
, http://www.100md.com +
TTCTGCAGGACGGCGTGAACTATGC
E2
964—986
-
GTAGGATCCCAGTTCATCATCAT
E3
959—981
+
TGGAATTCGATGATGAACTGGTC
E4
1741—1763
, 百拇医药
-
TTTCTGCAGCAATCCGTGGGGCA
E5
1444—1467
+
CAGAAGCTTTATTGCTGGCACTAC
E6
2200—2223
-
TATCTGCAGCATCATCCACAAGCA
Nucleotide location:Site A of the first ATG on Chinese Hebei strain[2]recognized location 1.GAATTC:EcoRI recognition sequence. GGATCC:BamH I recognition sequence. CTGCAG:Pst I recognition sequence. AAGCTT:HindⅢ recognition sequence.结 果
, http://www.100md.com
一、RT-PCR 产物克隆

Fig 1 Fragmental clonig of PCR products.
M: SppI/EcoRI DNA maker
1. linear pHC780;2. pHC780/HindIII+PstI;
3. linear pHC805;4. pHC805/EcoRI +PstI;
5. linear pHC 528;6. pHC528/BamH I+PstI;
7. linear pHC578;8. pHC578/EcoRI+BamHI;
, 百拇医药
9. linear pHC324;10. pHC324/EcoRI+BamHI;
λ. λDNA/EcoRI +BamHI.
图1 PCR产物分段克隆
NC1 和NC2 引物扩增的产物长324 bp,钝端正向插入pUC18的Sma I 切点重组为pHC324。C1和C2 引物扩增的产物长578 bp,粘端反向插入pUC18的EcoR I和BamH I双切点重组为pHC578。E1和E2引物扩增的产物长528 bp,粘端正向插入pUC18的BamH I和Pst I双切点重组为pHC528。E3和E4引物扩增的产物长805 bp,粘端反向插入pUC18的EcoRI 和Pst I双切点重组为pHC805。E5和E6引物扩增的产物长780 bp,粘端正向插入pUC18的HindⅢ和PstI双切点重组为pHC780(图1)。
, 百拇医药 二、第一步拼接克隆
分别以EcoRI与BamHI双切pHC578和以Pst I与BamH I双切pHC528,切出的克隆基因以连接PCR拼接后粘端反向插入pUC18的EcoRI 和BamHI双切点重组为pHC1050。分别以EcoRI与PstI双切pHC805 和以HindⅢ与Pst I 双切pHC780,切出的克隆基因以连接PCR拼接后粘端反向插入pUC18的EcoRI 与Pst I 双切点重组为pHC1260(图2)。

Fig 2 Splicing cloning of PCR products.
1. linear pHC1050;2. pHC1050/EcoRI+BamHI;
3. linear pHC1260; 4. pHC1260/EcoRI+PstI;
, 百拇医药
5. linear pHC2294; 6. pHC2294/EcoRI+PstI;
7. linear pHC2547; 8. pHC2547/EcoRI +PstI;
M.SppI/EcoRI DNA Maker.
图2 PCR产物拼接克隆
三、第二步拼接克隆
分别以EcoRI与BamHI 双切pHC1050和以EcoRI与Pst I双切pHC 1260,切出的克隆基因以连接PCR拼接后粘端反向插入pUC18 的EcoRI与Pst I双切点重组为pHC2294(图2),pHC2294含HCV几乎全部的结构蛋白编码序列。
四、第三步拼接克隆
, 百拇医药
以EcoRI与Pst I 双切pHC2294和以EcoRI与BamHI双切pHC324,切出的克隆基因以连接PCR拼接后钝端正向插入pGEM-3Zf(+)的Sma I切点重组为pHC2547(图2),pHC2547含HCV全部5'NCR序列和几乎全部的结构蛋白编码序列。
五、pHC2547鉴定
图3可见,由324 bp、578 bp、528 bp、805 bp 和780 bp这5个片段拼接而来的目的基因电泳位置(第8 电泳道)略低于对照分子量标准(M泳道)的第6条带(2.7 kb ),与目的基因的理论长度2547 bp相符;pGEM-3Zf(+)为3199 bp,迁移位置(第7泳道)低于对照标准的第5条带(3.5 kb);当与目的基因重组后分子量增大,迁移位置上升至接近于对照标准的第3条带(6.0 kb),与pHC2547的理论长度5746 bp相符。在PGEM-3Zf(+)多克隆位点各有1个EcoR I、Xba I、BamH I和Sac I切点,在目的基因第1345 nt和2238 nt位置依次有1个BamH I和Sac I切点,故pHC2547可被EcoR I 和Xba I 切为2个片段,其大小1条相当于pGEM-3Zf(+)空载体,1条相当于完整的克隆目的基因(第5泳道 );BamH I 也切出两条带(第4泳道),1条位于1.4 kb 和1.15 kb标准带之间,另1条略低于4.8 kb,与理论计算值4541 bp和1205 bp相符;Sac I切出的两条带(第3 泳道),1条小于2.7 kb但大于1.9 kb,另1条相当于3.5 kb,与理论计算值2238 bp和3508 bp也相符。对pHC2547以NC1和E6引物作长片段PCR扩增,得产物分子量与目的基因相若(第2泳道);以NC1和E2引物扩增,产物迁移位置稍低于1.4 kb(第1泳道),与实际大小1300 bp相符。以C1/E4引物经PCR制备地高辛素标记的1834 bp的HCV-cDNA探针对图3电泳模式作Southern 转移杂交结果见(图4)。第3和第5泳道的2条带中都是分子量小者阳性而大者阴性,说明小者含有而大者不含有探针同源序列;其余泳道则与电泳模式一致。因此拼接基因得以鉴定。

, 百拇医药
Fig 3 Identification of pHC2547 clone.
M:SppI/EcoRI NDA maker;
1.product of pHC2547 DNA amplified by prime NC1 and E2(1300 bp);
2.product of pHC2547 NDA amplified by prime NC1 and E6(2547bp);
3.pHC2547/SacI; 4.pHC2547/BamHI;
5.pHC2547/EcoRI+XbaI; 6.linear pHC2547;
7.linear pGEM-3Zf(+);
, 百拇医药
8.splicing target gene(2547bp).
图3 pHC2547 克隆鉴定

Fig 4 Mapping of Fig3 by Southern hybridization.
图4 图3的Southern杂交结果
六、逆转录病毒重组体pLHC的构建及鉴定
HindⅢ酶切pLNSX为载体,PstI与EcoRI切出pHC2547中的克隆序列为目的基因,经Klenow酶补平未端后以T4 DNA连接酶连接,连接物转化DH5a钙化菌并接种Luria-Bertani培养基平板。挑取单个菌落过夜培养,提取质粒DNA作斑点杂交,筛选阳性克隆菌。共筛选162个菌落,其中29个阳性,DNA酶切,发现有9个克隆其插入片段大小正确,4个为正向,5个为反向。其中1个正向者命名为pLHC,其酶切鉴定结果示(图5)。与对照DNA marker(M泳道)相比,目的基因(8泳道)略小于2.7 kb,载体(7泳道)约为6.0 kb,两者重组后(6泳道)大于8.0 kb,与实际大小2561 bp、6149 bp和8715 bp相符。以DNASIS软件模拟pLHC重组可见:Pst I与EcoR I切出目的基因补平末端后长2561 bp,上游残留pGEM 3Zf(+)的18 bp,下游4 nt则被Pst I切除;与pLNSX重组后长8715 bp,其中3167-5709 nt为HCV序列,3149-3166 nt为pGEM 3Zf(+)残留。模拟酶切提示:6050 nt有XbaI切点,3859 nt和5718 nt有Cla I切点,2798 nt和4507 nt有BamH I切点,1132 nt、1140 nt、1316 nt、1838 nt、2761 nt和7969 nt有Pst I切点,2 nt、1615 nt有EcoR I切点。酶切pLHC结果Xba I使之线性化(6泳道),Cla I切出6823 bp和1860 bp 2个片段(5泳道),BamH I切出6974 bp和1709 bp两个片段(4泳道),Pst I切出5208 bp和1846 bp、923 bp、522 bp和276 bp 5个片段(3泳道),EcoR I切出7070 bp和1613 bp 2个片段(2泳道)。而资作对照的EcoR I切空载体则切出4536 bp和1613 bp 2个片段(1泳道).实际结果与计算机推导完全一致。 (图6) 为pLHC的PCR鉴定结果,引物见前文[2],都扩增出预期大小的产物。

, http://www.100md.com
Fig 5 Identifieation of pLHC by enzymes.
M. SppI/EcoR I DNA marker;
1. pLNSX/EcoR I; 2. pLHC/EcoR I;
3. pLHC/Pst I;4. pLHC/BamH I;
5. pLHC/Cla I; 6. pLHC/Xba I;
7. pLNSX/Hind III; 8. pHC2547/EcoR I +Pst I.
图5 pLHC酶切鉴定

, 百拇医药
Fig 6 Identification of pLHC DNA by PCR.
M. SppI/EcoRI DNA maker;
1.product amplified by Prime NC1 and E6(2547bp);
2.product amplified by Prime E3 and E6(1265bp);
3.product amplified by Prime NC3 and E2(1260bp);
4.product amplified by prime C1 and E6(2294bp);
5.product amplified by prime NC1 and E2(1310bp);
, 百拇医药
6.product amplified by prime E1 and E2 (528bp);
7.product amplified by prime C1 and C2(578bp);
8.product amplified by prime NC1 and NC2(324bp).
图6 pLHC DNA的PCR鉴定
讨 论
HCV是第一个以反向生物学方法阐明的人类病毒。其病毒颗粒虽未确认,但基因序列却已克隆。HCV全基因组长9600 nt,携一个开放读框(open reading frame,ORF),后者又划分为若干基因区。由5′→3′方向排序依次为C、E1、E2/NS1、NS2-NS5共7个区。其中C区编码病毒的核壳蛋白,E1区编码病毒的包膜蛋白,E2/NS1区则可能编码HCV的第二种包膜蛋白,这3个区包含HCV的全部结构蛋白编码基因[1]。ORF上游还有一个保守的非编码区,称为5′NCR,这是调控HCV基因组复制和表达的关键序列[4]。
, http://www.100md.com
HCV 基因表达已有成熟的结果。重组的HCV 核壳蛋白一直以来都是ELISA 检测血清抗HCV 免疫诊断试剂的关键抗原,其来源包括原核表达、酵母表达[5]、昆虫细胞表达[6]和哺乳动物细胞表达[7]。重组E蛋白曾是研究HCV基因重组疫苗的热点,尽管最终归于失败,但却以此帮助了解到,HCV 具有准种系病毒(QUASISPECIES)这一新的特点[8]。有关非结构蛋白基因的表达也有不少报道。HCV的5′NCR序列对HCV基因复制和表达有极其重要的调控作用,它不仅含有病毒基因复制所需的核糖体进入位点,而且还有其它一些重要的顺式调控元件[4]。体外表达实验中,反义寡核酸选择性的封阻其某一区域可强力抑制后续序列的基因表达,但还缺乏细胞内和体内的实验依据[9]。
逆转录病毒载体能有效介导外源基因整合至宿主细胞染色体中,这一效率高达100%,介入的基因也几乎均能获得有效表达。此优点使逆转录病毒载体成为介导人体基因治疗的重要手段之一[10]。本文为研究HCV 的NCR序列对其结构蛋白编码基因表达的调控作用,拼接重组了包括完整5′NCR和全部结构蛋白编码基因的连续序列作为目的基因,顺向插入逆转录病毒载体pLNSX的SV40启动子下游,构建了pLHC重组体,这为进一步研究细胞内基因表达调控和转基因动物及基因治疗提供了可靠的物质基础。
, http://www.100md.com
[基金项目] 国家自然科学基金资助(No.39570285 )
Tel: 020-87333619; E-mail:kycao@gzsums. edu.cn
[参 考 文 献]
[1] 金冬雁. 丙型肝炎病毒分子生物学研究动态和发展趣向[M]. 见:侯云德,金冬雁,主编. 现代分子病毒学选论. 北京:科学出版社, 1994. 244-260.
[2] 毕胜利,白宪鹤,刘崇柏,等.我国丙型肝炎病毒河北株基因组的克隆及cDNA全序列的测序与分析[J]. 中华实验和临床病毒学杂志,1992,6(4):425-434.
[3] 吕 凌,王 斌. HCV 5′NCR 序列克隆和转录重组质粒构建[J]. 中山医科大学学报,1996,17(3):166-170
, 百拇医药
[4] Fukushi S, Katayama K, Kurihara C, et al. Complete 5’ noncoding region is necessary for the efficient internal initiation of hepatitis C virus RNA[J] . Biochem Biophys Res Commun,1994,199(2):425-432.
[5] Fillice G, Patruno S, Campisi D , et al. Specificity and sensitivity of 3rd generation EIA for detection of HCV antibodies among introvenous drug-users[J]. Microbiologica,1993,16(1):35-42.
[6] Hirowatari Y, Hijikata M, Tanji Y , et al . Expression and processing of putative nonstructural proteins of hepatitis C virus in insect cells using baculovirus vector[J]. Virus Res ,1995,35(1):43-61.
, 百拇医药
[7] Chang SC, Yen JH , Kang HY,et al . Nuclear localization signals in the core protein of hepatitis C virus[J]. Biochem Biophys Res Commun , 1994,205(2):1284-1290.
[8] Bukh J, Miller RH, Purcell RH. Genetic heterogeneity of hepatitis C virus: quasispecies and genotypes[J]. Semin liver Dis, 1995,15(1):41-63.
[9] Wakiba T, Wands JR. Specific inhibition of hepatitis C virus expression by antisense oligonucleotides[J]. J Bio Chem, 1994, 269(9): 14205-14210.
[10] Aoki K,Yochida T, Sugimura T, et al. Liposome-mediated in vivo gene transfer of antisense K-ras construct inhibits pancreatic tumor dissemination in the murine peritoneal cavity[J]. Cancer Res, 1995,55(17): 3810-3816.
[收稿日期] 2000-04-17 [修回日期] 2000-07-20, 百拇医药