DNA 芯片——逆向点杂交技术
作者:郭葆玉
单位:郭葆玉(第二军医大学药学院生化药学教研室, 上海 200433.)
关键词:DNA芯片;逆向点杂交
第二军医大学学报000827 [中图分类号] R 789 [文献标识码]
[文章编号] 0258-879X(2000)08-0789-02
自1995年斯坦福大学的Schena M 和Brown PO 等[1] 发表第1篇基因表达阵列芯片以来,国内外兴起了一股微点阵(microarrays)技术——DNA芯片大浪潮。DNA芯片技术的开发与应用已在世纪末为研究AIDS、癌症过程、某些疾病诊断、基因治疗、新食品开发、新药研制及生命科学开辟了一条全新的璀璨大道。虽然,目前就预言芯片技术对于生命科学所造成的冲击可能为时尚早。但是,可以展望在不久的将来,DNA芯片技术可以取代DNA自动化测序,广泛应用于新药研制和代替PCR技术对疾病做出诊断。从这个意义上说,微点阵技术完全可以与单克隆抗体(McAb)技术、PCR技术、重组DNA技术相媲美,从而成为21世纪生命科学研究的主要手段。
, 百拇医药
1 高通量、高并行、高精度的DNA芯片
简言之,芯片就是利用微点阵技术将大量的各种探针整齐而有序地排列在一小片如指甲盖大小的固相基质(solid phase)如玻片、尼龙膜等上面。使研究者能就一个问题去探讨造成该问题的最初成因、影响事件的途径(pathway links)、事件的发展过程(processing stage)和控制的制导机制(regulation mechanism),进而能以居高临下的“宏观”方式进行俯视,做出判断,提出对策。故此,就如同存储大量信息的电脑芯片一样,利用微点阵技术可将成千上万的生物密码资料储存在一片固相基质上,这个片子被称作DNA芯片。DNA芯片技术是集当今生物科技进步之大成,可将研究信息资料的点、线、面整体呈现在立体多维空间中进行高通量的快速分析,得到高精度大信息量结果。而以往的实验方式只能针对某一独立个体或个别简单单元进行分析,很难就复杂交错的多重生物反应的过程及丝丝紧扣、盘根错节的交互作用(cascade)给予研究者一个清晰完全的信息。
, 百拇医药
2 反向点杂交——DNA芯片的原理
Southern S等在20多年前,首次利用标记的核酸探针(labeled nucleic acid probe)通过分子杂交来探测固定在滤膜上受测样本的核酸序列。通过同源DNA分子之间的互补特性,经由标记探针杂交的位点可以很容易地发现具有互补序列的核酸的存在,进而演绎推论实验结果。Southern blot或Northern blot是将受测样本固定在纤维膜或尼龙膜上,再利用特异的标记探针来检测样品中是否存在互补的核酸序列。DNA芯片虽然在原理上与其相似,但却巧妙地反其道而用之,即反方向的将各种探针固定在基质上,用以检测受检的各种标记样品中与探针互补的核酸物质的变化。“规范”的DNA芯片技术是将探针,DNA或cDNA,以高密度点阵固定排列在经处理过的载玻片或尼龙膜的表面上,而底物则是标记了荧光或核素的能与其互补的靶核酸——DNA,cDNA或RNA。样本中的靶核酸,会杂交在微点阵上含互补核酸序列的探针样点上,再经洗涤,将没有杂交的游离样本核酸去除,就可纪录下有杂交反应样点的位置。只需1次实验,cDNA微点阵就能够将成千上万欲研究的表达基因记录下来(图1)。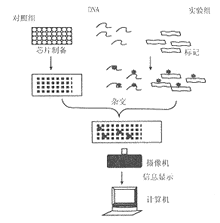
, 百拇医药
图 1 cDNA微点阵流程示意图
3 DNA芯片的类型
将DNA探针固定到芯片上的方法有多种,但归纳起来有两种,一种是光引导原位合成法(light-directed synthesis),还有一种是电压打印原位合成法。第1种方法类似于照相平板技术在半导体底面绝缘板上制作微型印刷线路的原理,只不过芯片制作中首先将支持物羟基化后再用光敏基因对支持物进行保护,选用罩光膜在需聚合部位透光,其余部位遮光。这样,当一定的光亮度照射支持物时,部分羟基基因解除保护。由于单体分子一端解除保护活化,而另一端仍受光敏基因保护,故发生偶联的部分在基因反应后仍带有光敏部分基因。这样通过控制罩光膜图案可使一些区域活化,如此可将已设定的探针在支持物上实现有序反应而制备完成。据介绍,这种方法可在8 h内完成48的探针合成,与传统方法相比其效率是不可想象的。第二种电压打印法如同平时电脑上连接的喷墨打印机的原理,即将墨盒中的墨汁用GACT 4种碱基替代,通过计算机控制的合成的DNA探针喷洒预设区域,一般合成40~50个碱基的短链后高效、正确地在支持物上进行阵列固定,上述方法由美国Affymtrix发明。
, 百拇医药
4 标本标记与杂交
标本标记主要利用荧光素、Cy3,Cy5以及放射性核素
32 P,33P,35S,125I,3H等。一般来说,荧光素标记能避免核素的污染,安全可靠,核素中35S的半衰期较长(3~6个月),较为理想且敏感性高,目前大多数实验室多选择它作为标记物。杂交条件尚需摸索,特别是洗膜条件需经多次试验才能得出较好的方案。例如,一个来源的标本总mRNA分别和1∶10 000的已知量mRNA(一般为乙酰胆碱受体AChR的mRNA)为内参照,做第一次反转录,各自的荧光素和丽丝胺核苷酸类似物标记,然后标记物等比例混合,再与芯片进行杂交洗膜,光电倍增信号CCD照相机摄取图像信息资料,计算机分析后即可提供数据。值得注意的是,应排除假阳性,特别是敏感性越高,假阳性就越大,这就要求有实验和经验来避免上述可能性的发生。
, 百拇医药
5 DNA芯片广阔的应用前景
迄今,全世界不同生物基因的研究仍在持续进行中,其中已有包括啤酒酵母在内的17种生物基因被基因物理图谱及测序完全破译。而人类30亿个核苷酸的基因测序也预期在公元2000~2003年全部完成。虽然在人类的基因中已经被编排有超过110万个表达序列靶位点,控制着52 907个人类基因的表达,然而对于80%以上的这些基因,它们有什么功能,如何表达,如何被调节,至今仍不清楚。DNA芯片是一个非常有潜力的芝麻,它能够为科学家开启一扇了解基因的结构与功能——人类后基因组工程的新窗口。DNA芯片亦可用在分析基因的表达上,即生物体中的mRNA是否被诱导产生表达或表达抑制或表达丧失的种类及程度,了解更为完整的转录作用和基因调控的变化过程。DNA芯片还可用于生物的基因表型(genotyping)的分类[2],可从上百个样本中的上万个基因座(loci)中探查出等位基因(alleles)的异同,进而提供信息用作研究繁杂的多基因异常引起的疾病。目前已广泛应用于后基因组的研究有急性白血病[3]、黑色素病[4]、卵巢上皮癌[5]、乳腺癌[6]、肾癌[7]、前列腺癌[8]、横纹肌肉瘤[9]等的发生分子生物学。也可用于细胞因子、粘附分子、受体、离子通道、信号转导、细胞周期、应激反应、修复、转导翻译的调控、蛋白质化学、酶、持家基因等的研究,以及凋亡蛋白、霍奇金病细胞亚群的鉴定等所谓发散性大细胞淋巴瘤特征型鉴定(DLBCL),这无疑对癌症诊断及发生机制的认识提供了有力的工具。
, 百拇医药
DNA芯片也可对个体和家族群体、不同人种和不同生物种群中等位基因的多态性(polymorphism)进行分析来控制遗传性疾病。由于一片DNA芯片上含有成千上万个基因样点,并且目前的方法也能精确检测到极细微的细胞内膜的变化,乃至一个细胞内少数几个mRNA上的甚至一个碱基的变化,从而使研究者能快速通过计算机处理过的数据得到非常完整、精确的信息资料,这无疑对人类认识自身、认识世界和控制疾病等具有十分重要的意义。同时,DNA芯片也是唯一可提供大量资料用于丰度极低或不易获取样品的研究途径。目前,以核酸为样点(material matrix)的DNA芯片已在广泛蓬勃的发展中,尤其在基因表达、基因作图(mapping)、疾病诊断、新药筛选、靶点寻找、药效评价等方面有非常显著的用途,某些甚至可以做测序及检测细微核苷酸异常的芯片已经上市。以蛋白质、抗原、抗体、生物膜、各种配体、受体及细胞内成分为样点的生物芯片亦是人们颇感兴趣的研究方向和热点,其价值将不可估量。可以预言,在不久的将来,更成熟、灵敏的DNA芯片技术可提供给生命科学的研究,用以了解和破译生物的生命密码的全部信息。
, 百拇医药
[作者简介] 郭葆玉(1951-), 男(汉族), 博士, 教授,博士生导师.
[参 考 文 献]
[1] Schena M, Shalon D, Davis RW, et al. Quantitative monitoring of gene expression patterns with a complementary DNA microarray[J] . Science, 1995, 270(5235):467-470.
[2] Schena M, Shalon D, Heller R et al. Parallel human genome analysis:microarray-based expression monitoring of 1000 genes[J]. PNAS. USA, 1996, 198(12):10614-10619.
, 百拇医药
[3] Golub TR, Slonim DK, Tamayo P, et al . Molecular classification of cancer: class discovery and class prediction by gene expression monitoring[J]. Science, 1999, 286(5439):531-537.
[4] Derisi J, Penland L, Brown PO, et al. Use of a cDNA microarray to analyse gene expression patterns in human cancer[J]. Nat Genet, 1996, 14(4):457-460.
[5] Chuaqui RF, Cole KA, Emmert-Buck MR, et al. Histopathology and molecular biology of ovarian epithelial tumors[J]. Ann Diagn Pathol, 1998, 2(3):195-207.
, 百拇医药
[6] Osin P, Schipley J, Lu YJ, et al. Experimental pathology and breast cancer genetics:new technologics[J]. Recent Results Cancer Res, 1998, 152:35-48.
[7] Moch H, Schraml P, Bubendorf L, et al. High throughput tissue microarray analysis to evaluate genes uncovered by cDNA microarray screening in renal cell carcinoma[J]. Am J Pathol, 1999, 154(4):981-986.
[8] Bubendorf L, Kolmer M, Kononen J, et al. Hormone therapy failure in human prostate cancer:analysis by complementary DNA and tissue microarrays[J]. J Natl Cancer Inst, 1999, 91(20):1758-1764.
[9] Khan J, Simon R, Bittner M, et al. Gene expression profiling of alveolar rhabdomyosarcoma with cDNA microarrays[J]. Cancer Res, 1998, 58(22):5009-5013.
[收稿日期] 2000-06-01
[修回日期] 2000-07-21, http://www.100md.com
单位:郭葆玉(第二军医大学药学院生化药学教研室, 上海 200433.)
关键词:DNA芯片;逆向点杂交
第二军医大学学报000827 [中图分类号] R 789 [文献标识码]
[文章编号] 0258-879X(2000)08-0789-02
自1995年斯坦福大学的Schena M 和Brown PO 等[1] 发表第1篇基因表达阵列芯片以来,国内外兴起了一股微点阵(microarrays)技术——DNA芯片大浪潮。DNA芯片技术的开发与应用已在世纪末为研究AIDS、癌症过程、某些疾病诊断、基因治疗、新食品开发、新药研制及生命科学开辟了一条全新的璀璨大道。虽然,目前就预言芯片技术对于生命科学所造成的冲击可能为时尚早。但是,可以展望在不久的将来,DNA芯片技术可以取代DNA自动化测序,广泛应用于新药研制和代替PCR技术对疾病做出诊断。从这个意义上说,微点阵技术完全可以与单克隆抗体(McAb)技术、PCR技术、重组DNA技术相媲美,从而成为21世纪生命科学研究的主要手段。
, 百拇医药
1 高通量、高并行、高精度的DNA芯片
简言之,芯片就是利用微点阵技术将大量的各种探针整齐而有序地排列在一小片如指甲盖大小的固相基质(solid phase)如玻片、尼龙膜等上面。使研究者能就一个问题去探讨造成该问题的最初成因、影响事件的途径(pathway links)、事件的发展过程(processing stage)和控制的制导机制(regulation mechanism),进而能以居高临下的“宏观”方式进行俯视,做出判断,提出对策。故此,就如同存储大量信息的电脑芯片一样,利用微点阵技术可将成千上万的生物密码资料储存在一片固相基质上,这个片子被称作DNA芯片。DNA芯片技术是集当今生物科技进步之大成,可将研究信息资料的点、线、面整体呈现在立体多维空间中进行高通量的快速分析,得到高精度大信息量结果。而以往的实验方式只能针对某一独立个体或个别简单单元进行分析,很难就复杂交错的多重生物反应的过程及丝丝紧扣、盘根错节的交互作用(cascade)给予研究者一个清晰完全的信息。
, 百拇医药
2 反向点杂交——DNA芯片的原理
Southern S等在20多年前,首次利用标记的核酸探针(labeled nucleic acid probe)通过分子杂交来探测固定在滤膜上受测样本的核酸序列。通过同源DNA分子之间的互补特性,经由标记探针杂交的位点可以很容易地发现具有互补序列的核酸的存在,进而演绎推论实验结果。Southern blot或Northern blot是将受测样本固定在纤维膜或尼龙膜上,再利用特异的标记探针来检测样品中是否存在互补的核酸序列。DNA芯片虽然在原理上与其相似,但却巧妙地反其道而用之,即反方向的将各种探针固定在基质上,用以检测受检的各种标记样品中与探针互补的核酸物质的变化。“规范”的DNA芯片技术是将探针,DNA或cDNA,以高密度点阵固定排列在经处理过的载玻片或尼龙膜的表面上,而底物则是标记了荧光或核素的能与其互补的靶核酸——DNA,cDNA或RNA。样本中的靶核酸,会杂交在微点阵上含互补核酸序列的探针样点上,再经洗涤,将没有杂交的游离样本核酸去除,就可纪录下有杂交反应样点的位置。只需1次实验,cDNA微点阵就能够将成千上万欲研究的表达基因记录下来(图1)。

, 百拇医药
图 1 cDNA微点阵流程示意图
3 DNA芯片的类型
将DNA探针固定到芯片上的方法有多种,但归纳起来有两种,一种是光引导原位合成法(light-directed synthesis),还有一种是电压打印原位合成法。第1种方法类似于照相平板技术在半导体底面绝缘板上制作微型印刷线路的原理,只不过芯片制作中首先将支持物羟基化后再用光敏基因对支持物进行保护,选用罩光膜在需聚合部位透光,其余部位遮光。这样,当一定的光亮度照射支持物时,部分羟基基因解除保护。由于单体分子一端解除保护活化,而另一端仍受光敏基因保护,故发生偶联的部分在基因反应后仍带有光敏部分基因。这样通过控制罩光膜图案可使一些区域活化,如此可将已设定的探针在支持物上实现有序反应而制备完成。据介绍,这种方法可在8 h内完成48的探针合成,与传统方法相比其效率是不可想象的。第二种电压打印法如同平时电脑上连接的喷墨打印机的原理,即将墨盒中的墨汁用GACT 4种碱基替代,通过计算机控制的合成的DNA探针喷洒预设区域,一般合成40~50个碱基的短链后高效、正确地在支持物上进行阵列固定,上述方法由美国Affymtrix发明。
, 百拇医药
4 标本标记与杂交
标本标记主要利用荧光素、Cy3,Cy5以及放射性核素
32 P,33P,35S,125I,3H等。一般来说,荧光素标记能避免核素的污染,安全可靠,核素中35S的半衰期较长(3~6个月),较为理想且敏感性高,目前大多数实验室多选择它作为标记物。杂交条件尚需摸索,特别是洗膜条件需经多次试验才能得出较好的方案。例如,一个来源的标本总mRNA分别和1∶10 000的已知量mRNA(一般为乙酰胆碱受体AChR的mRNA)为内参照,做第一次反转录,各自的荧光素和丽丝胺核苷酸类似物标记,然后标记物等比例混合,再与芯片进行杂交洗膜,光电倍增信号CCD照相机摄取图像信息资料,计算机分析后即可提供数据。值得注意的是,应排除假阳性,特别是敏感性越高,假阳性就越大,这就要求有实验和经验来避免上述可能性的发生。
, 百拇医药
5 DNA芯片广阔的应用前景
迄今,全世界不同生物基因的研究仍在持续进行中,其中已有包括啤酒酵母在内的17种生物基因被基因物理图谱及测序完全破译。而人类30亿个核苷酸的基因测序也预期在公元2000~2003年全部完成。虽然在人类的基因中已经被编排有超过110万个表达序列靶位点,控制着52 907个人类基因的表达,然而对于80%以上的这些基因,它们有什么功能,如何表达,如何被调节,至今仍不清楚。DNA芯片是一个非常有潜力的芝麻,它能够为科学家开启一扇了解基因的结构与功能——人类后基因组工程的新窗口。DNA芯片亦可用在分析基因的表达上,即生物体中的mRNA是否被诱导产生表达或表达抑制或表达丧失的种类及程度,了解更为完整的转录作用和基因调控的变化过程。DNA芯片还可用于生物的基因表型(genotyping)的分类[2],可从上百个样本中的上万个基因座(loci)中探查出等位基因(alleles)的异同,进而提供信息用作研究繁杂的多基因异常引起的疾病。目前已广泛应用于后基因组的研究有急性白血病[3]、黑色素病[4]、卵巢上皮癌[5]、乳腺癌[6]、肾癌[7]、前列腺癌[8]、横纹肌肉瘤[9]等的发生分子生物学。也可用于细胞因子、粘附分子、受体、离子通道、信号转导、细胞周期、应激反应、修复、转导翻译的调控、蛋白质化学、酶、持家基因等的研究,以及凋亡蛋白、霍奇金病细胞亚群的鉴定等所谓发散性大细胞淋巴瘤特征型鉴定(DLBCL),这无疑对癌症诊断及发生机制的认识提供了有力的工具。
, 百拇医药
DNA芯片也可对个体和家族群体、不同人种和不同生物种群中等位基因的多态性(polymorphism)进行分析来控制遗传性疾病。由于一片DNA芯片上含有成千上万个基因样点,并且目前的方法也能精确检测到极细微的细胞内膜的变化,乃至一个细胞内少数几个mRNA上的甚至一个碱基的变化,从而使研究者能快速通过计算机处理过的数据得到非常完整、精确的信息资料,这无疑对人类认识自身、认识世界和控制疾病等具有十分重要的意义。同时,DNA芯片也是唯一可提供大量资料用于丰度极低或不易获取样品的研究途径。目前,以核酸为样点(material matrix)的DNA芯片已在广泛蓬勃的发展中,尤其在基因表达、基因作图(mapping)、疾病诊断、新药筛选、靶点寻找、药效评价等方面有非常显著的用途,某些甚至可以做测序及检测细微核苷酸异常的芯片已经上市。以蛋白质、抗原、抗体、生物膜、各种配体、受体及细胞内成分为样点的生物芯片亦是人们颇感兴趣的研究方向和热点,其价值将不可估量。可以预言,在不久的将来,更成熟、灵敏的DNA芯片技术可提供给生命科学的研究,用以了解和破译生物的生命密码的全部信息。
, 百拇医药
[作者简介] 郭葆玉(1951-), 男(汉族), 博士, 教授,博士生导师.
[参 考 文 献]
[1] Schena M, Shalon D, Davis RW, et al. Quantitative monitoring of gene expression patterns with a complementary DNA microarray[J] . Science, 1995, 270(5235):467-470.
[2] Schena M, Shalon D, Heller R et al. Parallel human genome analysis:microarray-based expression monitoring of 1000 genes[J]. PNAS. USA, 1996, 198(12):10614-10619.
, 百拇医药
[3] Golub TR, Slonim DK, Tamayo P, et al . Molecular classification of cancer: class discovery and class prediction by gene expression monitoring[J]. Science, 1999, 286(5439):531-537.
[4] Derisi J, Penland L, Brown PO, et al. Use of a cDNA microarray to analyse gene expression patterns in human cancer[J]. Nat Genet, 1996, 14(4):457-460.
[5] Chuaqui RF, Cole KA, Emmert-Buck MR, et al. Histopathology and molecular biology of ovarian epithelial tumors[J]. Ann Diagn Pathol, 1998, 2(3):195-207.
, 百拇医药
[6] Osin P, Schipley J, Lu YJ, et al. Experimental pathology and breast cancer genetics:new technologics[J]. Recent Results Cancer Res, 1998, 152:35-48.
[7] Moch H, Schraml P, Bubendorf L, et al. High throughput tissue microarray analysis to evaluate genes uncovered by cDNA microarray screening in renal cell carcinoma[J]. Am J Pathol, 1999, 154(4):981-986.
[8] Bubendorf L, Kolmer M, Kononen J, et al. Hormone therapy failure in human prostate cancer:analysis by complementary DNA and tissue microarrays[J]. J Natl Cancer Inst, 1999, 91(20):1758-1764.
[9] Khan J, Simon R, Bittner M, et al. Gene expression profiling of alveolar rhabdomyosarcoma with cDNA microarrays[J]. Cancer Res, 1998, 58(22):5009-5013.
[收稿日期] 2000-06-01
[修回日期] 2000-07-21, http://www.100md.com